Revisión clínica
← vista completaPublicado el 26 de septiembre de 2020 | http://doi.org/10.5867/medwave.2020.08.8037
Origen, características estructurales, medidas de prevención, diagnóstico y fármacos potenciales para prevenir y controlar COVID-19
Origin, structural characteristics, prevention measures, diagnosis and potential drugs to prevent and COVID-19
Resumen
El síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2) es responsable de la enfermedad denominada COVID-19 (acrónimo del inglés Coronavirus Disease-2019). Esta enfermedad fue detectada inicialmente en la ciudad de Wuhan, China en diciembre de 2019. Las personas contagiadas con COVID-19 presentan síntomas variados, dependiendo de su estado de salud y edad. Los síntomas más comunes son fiebre, tos, mialgia, fatiga, odinofagia y disnea. También se ha observado que en algunos pacientes, la infección es asintomática. Los adultos mayores de 60 años infectados son el grupo de pacientes más susceptibles a desarrollar estados severos de COVID-19 y se presenta comorbilidad en presencia de enfermedades crónicas. Por otra parte, también es importante disponer de pruebas que permitan detectar al SARS-COV-2 y seguir la evolución de COVID-19 de forma rápida, confiable y barata. Para lograr esto, existen pruebas de reacción en cadena de la polimerasa de transcripción inversa en tiempo real (RT-PCR), de amplificación isotérmica de ácido nucleico y de inmunoestimulación enzimática. Actualmente, no existen tratamientos para la prevención del contagio y combatir los efectos del virus en la salud humana. Sin embargo, en el mundo hay grupos de investigación que están realizando pruebas in vitro, in vivo e in silico para encontrar fármacos que sean capaces de prevenir y/o controlar la infección en humanos con SARS-CoV-2. La cloroquina, hidroxicloroquina, remdesivir, interferon-2b y oseltamivir son algunas de las opciones farmacológicas que están siendo evaluadas en pruebas clínicas para la profilaxis de COVID-19. El objetivo de la presente revisión consiste en establecer un marco de referencia de la clasificación taxonómica del SARS-CoV-2 y la relación que guardan con otros coronavirus, así como su estructura y forma de propagarse en el ser humano. También se presentan las características y síntomas de pacientes con COVID-19, los métodos de detección y potenciales tratamientos.
Ideas clave
- A partir de diciembre de 2019, el SARS-CoV-2 se propagó desde la ciudad de Wuhan en China a prácticamente todas las ciudades del mundo, incluyendo las de Latinoamérica, provocando un problema sanitario a nivel global por ser una zoonosis sin precedentes y desconocida. Ello ha marcado la aparición de diversas fuentes de información que se actualizan a cada momento.
- Debido a lo desconocido del tema, es importante contar con un panorama general sobre el origen y las características estructurales del SARS-CoV-2, el diagnóstico clínico y potenciales tratamientos para COVID-19, necesidad resuelta en parte por este artículo.
- La revisión bibliográfica está limitada a las publicaciones correspondientes hasta el mes de julio de 2020.
Introducción
Actualmente los avances tecnológicos han generado una conectividad nunca antes vista, lo que ha traído consigo no solo mejoras en nuestras condiciones de vida, sino también la aparición de nuevas enfermedades y su esparcimiento, como es el caso de la enfermedad por coronavirus (COVID-19, del inglés Coronavirus Disease) ocasionada por el SARS-CoV-2[1]. Este virus tiene similitud al SARS-CoV y MERS-CoV (MERS, acrónimo del inglés Middle East Respiratory Syndrome), coronavirus causantes de epidemias en este siglo[2],[3].
El SARS-CoV-2 a partir de diciembre de 2019 se propagó desde la ciudad de Wuhan en la provincia de Hubei, China a prácticamente todas las ciudades más importantes del mundo, incluyendo las de Latinoamérica. Esto ha provocado un verdadero problema sanitario a nivel global por ser una zoonosis sin precedentes y desconocida, lo que ha marcado la aparición de diversas fuentes de información que se actualizan momento a momento.
Las personas sintomáticas y asintomáticas de COVID-19 transmiten el SARS-CoV-2[4], y los pacientes jóvenes con una mediana de edad de 22 años (en un intervalo de 16 a 23 años) son los principales transmisores asintomáticos[5]. La transmisión se produce a través de las gotas generadas por la tos y los estornudos, fómites, y no se descartan otras vías como la fecal o la aérea[6]. Todo esto hace que el SARS-CoV-2 se propague fácilmente.
Por lo anterior, el presente artículo consiste en proporcionar un marco de referencia relacionado con la clasificación taxonómica, estructura y origen del SARS-CoV-2; así como los factores que promovieron su conversión en una pandemia. También se incluye información relacionada con el diagnóstico clínico y las pruebas que se están utilizando para la detección del SARS-CoV-2.
Finalmente, se presentan algunos estudios relacionados con fármacos con potencial uso para la prevención y tratamiento de COVID-19 a nivel in vitro, in vivo e in silico. Para cumplir con este objetivo, se llevó a cabo una revisión de revistas que de forma regular publican artículos sobre los coronavirus y, especialmente, en relación a SARS-CoV-2. En menor grado se revisaron páginas electrónicas de organismos internacionales, tales como la Organización Mundial de la Salud (OMS) y la Food and Drug Administration (FDA), entre otras.
Clasificación de los coronavirus
Los coronavirus pertenecen al orden de los Nidovirales, a la familia Coronaviridae y se clasifican según sus características genéticas preponderantes, que se localizan dentro de la poliproteína replicasa ORF1ab (pp1ab). Las características más distintivas de los coronavirus son:
- El tamaño del genoma de alrededor de 30 000 pares de bases, los coronavirus son los virus de ácido ribonucleico con los genomas más grandes. Esta capacidad de codificación expansiva parece proporcionar y necesitar una gran cantidad de estrategias de expresión génica.
- Expresión de muchos genes no estructurales por desplazamiento del marco ribosómico.
- Varias actividades enzimáticas únicas o inusuales codificadas dentro de la gran poliproteína replicasa-transcriptasa.
- Expresión de genes downstream por síntesis de ácido ribonucleico mensajero subgenómico anidado en 3', confiriéndole un gran parecido al ácido ribonucleico mensajero del hospedador.
El nombre del orden de los Nidovirales se deriva de estos ácidos ribonucleicos mensajeros 3' anidados. Las principales diferencias dentro de las familias de nidovirus están en el número, tipo y tamaño de las proteínas estructurales. Debido a que los virus tienden a mutar con bastante facilidad, se ha establecido que las cepas aisladas de distintas fuentes deben presentar más del 90% de homología (a nivel de aminoácidos) dentro de los dominios conservados de la replicasa para considerarse que son la misma especie[7]. A partir de estos criterios, los coronavirus de acuerdo al grupo de Estudio de Coronavirus del Comité Internacional de Taxonomía de Virus, se clasifican en cuatro géneros, divididos por agrupamientos filogenéticos, alfacoronavirus (acoronavirus), betacoronavirus (βcoronavirus), gammacoronavirus (γcoronavirus) y deltacoronavirus (δcoronavirus)[8]. Dentro del género βcoronavirus hay cuatro linajes, estos son los linajes A, B, C y D, Figura 1.
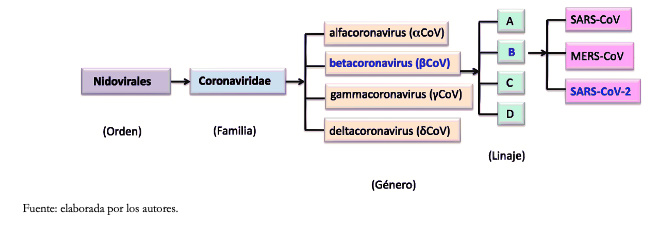 Tamaño completo
Tamaño completo Hay tres razones principales para la gran diversidad de coronavirus. En primer lugar, sus genomas son muy plásticos porque la diversidad baja de sus ácidos ribonucleicos polimerasas dependientes de ácido ribonucleico conduce a tasas altas de mutación de uno por 1000 a 10 000 nucleótidos replicados[9],[10]. En segundo lugar, los coronavirus tienen tasas altas de recombinación de ácido ribonucleico homólogo debido a su mecanismo único de cambio aleatorio de plantilla durante la replicación de ácido ribonucleico[11],[12]. En tercer lugar, los coronavirus tienen una capacidad mayor para incorporar nuevos genes respecto a otros virus de ácido ribonucleico, debido a sus genomas relativamente grandes (aproximadamente de 26 a 32 kilobases). Estos tres factores permiten generar diversas cepas y genotipos dentro de una sola especie de coronavirus. Además, las nuevas variantes o especies pueden ocasionar brotes zoonóticos.
Estructura de los viriones de coronavirus y mecanismo de acción del SARS-CoV-2
Los viriones de coronavirus contienen cuatro proteínas estructurales principales. Estas son las proteínas de espiga (S), membrana (M), envoltura (E) y nucleocápside (N). Todas están codificadas dentro del extremo 3' del genoma viral (Figura 2 y Tabla 1). Los coronavirus son aproximadamente esféricos y moderadamente pleomórficos. Por lo general, se ha reportado que los viriones tienen diámetros promedio de 80 a120 nanómetros. La proteína S (~ 150 kilodalton), utiliza una secuencia de señal N-terminal para obtener acceso al hospedero. Esta proteína constituye la estructura de espiga distintiva en la superficie del virus[13],[14]. Las espigas S, se proyectan a unos 17 a 20 nanómetros de la superficie del virión, con una base delgada que se hincha hasta un ancho de aproximadamente 10 nanómetros en la extremidad distal[15]. La proteína M es una glicoproteína estructural de membrana localizada en la envoltura del virión. Es pequeña (25 a 30 kilodalton), con tres dominios transmembrana[16] y desempeña un papel central en el ensamblaje del virus, convirtiendo las membranas celulares en fábricas donde el virus y los factores del hospedero se unen para producir nuevos viriones[17]. La proteína E (8 a 12 kilodalton) se encuentra en cantidades pequeñas dentro del virión. Las proteínas E de los coronavirus son muy variables, pero tienen una arquitectura común[18]. La proteína E tiene varias funciones, entre ellas facilitar el ensamblaje y la liberación del virus, y es necesaria para infectar nuevas células[19]. La proteína N constituye la única proteína presente en la nucleocápside. Se compone de dos dominios separados, un dominio N-terminal (NTD) y un dominio C-terminal (CTD), ambos capaces de unirse al ácido ribonucleico in vitro, pero cada dominio utiliza diferentes mecanismos para unirse al ácido ribonucleico. La unión óptima con el ácido ribonucleico requiere contribuciones de ambos dominios[20],[21]. Los βCoV exhiben un segundo tipo de proyección de superficie, de 5 a 10 nanómetros de largo debajo de las espigas S, constituida de una glucoproteína hemaglutinina-esterasa homodimérica, Figura 2. Esta proteína también es una mediadora de la unión al receptor. Los virus que expresan la proteína hemaglutinina-esterasa se unen a los eritrocitos y los aglutinan, y sólo se encuentra en los βcoronavirus de linaje A[22],[23].
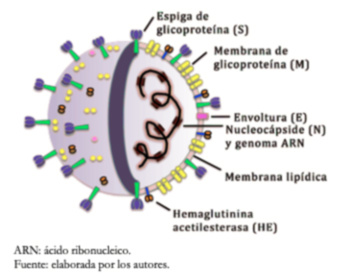 Tamaño completo
Tamaño completo En la mayoría de los coronavirus, la proteína S es escindida por una proteasa tipo furina de la célula hospedera en dos polipéptidos separados, denotados como S1 y S2[23]. S1 constituye el dominio de enlace al receptor de la proteína S permitiendo al virus penetrar la célula del hospedero, mientras que S2 contiene la maquinaria de fusión (Figura 3).
 Tamaño completo
Tamaño completo 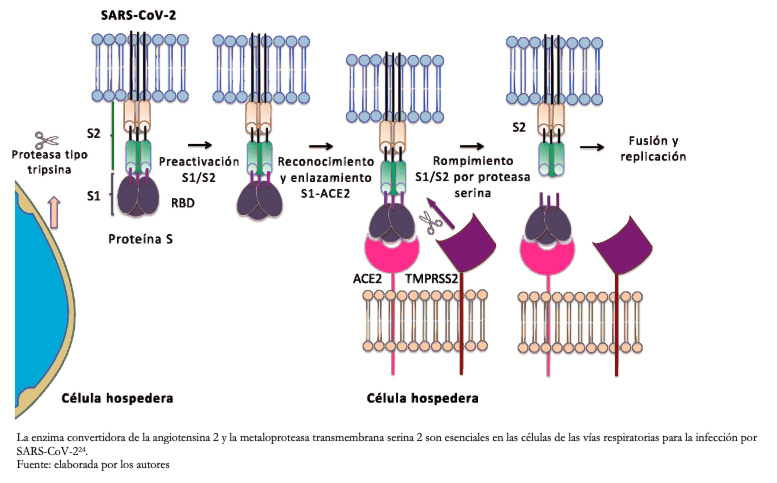 Tamaño completo
Tamaño completo Letko y Munster (2020) desarrollaron un enfoque para detectar rápidamente los βcoronavirus del linaje B, tal como el SARS-CoV y SARS-CoV-2, durante la invasión del receptor y su capacidad de infectar células de diferentes tipos de especies[25]. Este estudio evidenció que el procesamiento de la proteasa del hospedero durante la entrada viral es una barrera importante para varios βcoronavirus del linaje B y al eludir esta barrera, los virus ingresan a las células humanas a través de un receptor desconocido. También, los autores demostraron cómo los diferentes βcoronavirus del linaje B pueden recombinarse para ingresar a las células humanas y confirmar que la enzima convertidora de angiotensina 2 humana es el receptor del SARS-CoV-2.
La enzima convertidora de la angiotensina 2 está unida a la superficie externa de las membranas de células pulmonares, arteriales, de corazón, riñón e intestinos[26]. El fragmento S2 del SARS-CoV-2, es escindido por una serina proteasa de la superficie celular humana, metaloproteasa transmembrana serina 2 (Figura 3), que da como resultado la fusión de la membrana. De acuerdo con Hoffmann y colaboradores (2020), la enzima convertidora de la angiotensina 2 y la metaloproteasa transmembrana serina 2 son esenciales en las células de las vías respiratorias para la infección por SARS-CoV-2[24].
En relación a la enzima convertidora de la angiotensina 2 se conoce que se sobre expresa en pacientes con hipertensión, diabetes tipo 1 y 2 que han sido tratados con inhibidores de enzima convertidora de la angiotensina y bloqueadores del receptor angiotensina II tipo-1[27]. Esta condición puede generar en los pacientes con COVID-19 un curso clínico más severo que implica daños al miocardio, lo que ha generado especulación pues no está totalmente comprendida la asociación entre la sobreexpresión de enzima convertidora de la angiotensina 2 y el riesgo de infección con SARS-CoV-2 o la severidad de COVID-19[28].
Adicionalmente, para entender la relación que existe entre pacientes con enfermedades cardiovasculares y COVID-19, puede ser de gran utilidad el trabajo publicado por Chen y colaboradores (2020), quienes reportaron que de 99 pacientes confirmados con COVID-19 ingresados en el Hospital Wuhan Jinyintan, 13% presentaron creatina quinasa elevada y 76% mostraron una concentración elevada de lactato deshidrogenasa[29]. La creatina quinasa se encuentra prinpalmente en los músculos esqueléticos y miocardio, y en menor concentración en el cerebro. Una concentración alta de creatina quinasa puede indicar un daño en alguno de los músculos antes señalados. Al igual que la creatina quinasa, la lactato deshidrogenasa se utiliza para identificar lesiones en tejidos, entre ellos el miocardio; sin embargo, lactato deshidrogenasa es menos específica que creatina quinasa.
Un marcador muy sensible y específico de lesión del miocardio es la troponina I, la cual fue evaluada por Wang y colaboradores (2020a) en 138 pacientes hospitalizados con COVID-19 en el Hospital Zhongnan de la Universidad de Wuhan. Wang y colaboradores (2020) encontraron niveles de troponina I elevados en el 7,2% de los pacientes[30]. Cabe señalar que la disfunción de miocardio puede ser provocada indirectamente por COVID-19. Por ejemplo, inducida por un suministro reducido de oxígeno, insuficiencia pulmonar grave y una tormenta de citoquinas después de la infección por SARS-CoV-2. Sin embargo, también existe la posibilidad de que sea atribuible a la disminución de la actividad de enzima convertidora de la angiotensina 2 en el corazón. En la actualidad, todavía no es posible distinguir todos esos posibles promotores de la disfunción del miocardio.
El origen de los coronavirus que infectan a humanos
En tiempos relativamente recientes, al menos se han registrado cuatro eventos de saltos zoonóticos de coronavirus desde animales a humanos que han resultado en epidemias. El primer evento ocurrió con un β-coronavirus del linaje A, que involucró al menos nueve especies animales, incluyendo a humanos. Se especula que el salto zoonótico final de este βcoronavirus fue de bovinos a humanos hace unos 120 años, conduciendo al CoV OC43. Este virus junto con el HCoV-229E se aislaron e identificaron en los años 1967 y 1966, respectivamente. El CoV OC43 y el HCoV-229E fueron los primeros coronavirus identificados en humanos. Durante 40 años se pensó que estos coronavirus eran los únicos infectantes[31]. Estudios en voluntarios adultos sanos revelaron que la infección con estos dos coronavirus causa síntomas de resfriado común. Adicionalmente, en individuos infectados con HCoV-229E se presenta la rinitis, mientras que en pacientes infectados con HCoV-OC43 se presenta la odinofagia. Por lo tanto, estos dos virus no representan un problema severo de salud en el mundo.
El segundo evento de saltos zoonóticos ocurrió a principios del siglo XXI e involucró a un β-coronavirus del linaje B, dando origen al SARS-CoV humano en el año 2003. Este evento implicó a murciélagos, civetas de palmeras (Paguma larvata) y humanos, con el virus saltando de civetas de las palmeras a humanos[32]. La civeta de las palmeras es un alimento altamente apreciado en China. El SARS-CoV fue el responsable de la epidemia que dio inicio en Guandong, China en 2003[33]. Esta primera pandemia del siglo XXI generada por coronavirus humanos se controló ese mismo año implementando cuarentenas.
El tercer evento zoonótico ocurrió en el linaje C de los β-coronavirus, lo que resultó en la epidemia del síndrome respiratorio de Oriente Medio[34]. El MERS-CoV está estrechamente relacionado con el CoV HKU4 presente en el murciélago Tylonycteris (Ty-BatCoV HKU4) y el CoV HKU5 presente en el murciélago Pipistrellus (Pi-BatCoV HKU5), ambas especies originarias de Hong Kong[35]. Por otra parte, brotes de los virus HCoV-NL63 y HCoV-HKU1 aparecieron en los años 2004 y 2005, respectivamente. Estos últimos coronavirus humanos no son nuevos, simplemente no se habían detectado previamente. Adicionalmente, existen evidencias de que genotipos de la cepa HCoV-HKU1 se recombinaron de forma natural[36].
Las cuatro cepas de coronavirus humanos mencionadas anteriormente se clasifican como virus de resfriados comunes que en algunos casos afectan de manera severa a niños pequeños, adultos con enfermedades crónicas y ancianos. Otro β-coronavirus, de linaje C, fue el responsable de la infección respiratoria en humanos que generó el síndrome respiratorio del Medio Oriente en 2012 (MERS-CoV). En la actualidad este β-coronavirus está bien adaptado a los humanos[37]. Por lo tanto, a simple vista parece que los murciélagos son los hospederos naturales de los coronavirus que originaron el linaje C de β-coronavirus. Sin embargo, un número significativo de casos de contagios no estaban relacionados con la exposición directa a los murciélagos[38]. Un escenario más probable es que una sola variante de βcoronavirus en murciélagos saltó con éxito y se estableció rápidamente en una especie hospedera animal intermedia, por ejemplo, dromedarios y civetas, con la consecuente propagación incidental a la población humana. Tales eventos zoonóticos se facilitan a través de frecuentes interacciones hospedero intermedio-humano y, quizás, a través de adaptaciones virales adquiridas durante el salto inicial de la especie[35]. Por lo tanto, el intervalo de fuentes animales y las rutas de transmisión entre especies durante la generación del linaje C de β-coronavirus, todavía se desconocen.
El cuarto evento zoonótico corresponde al SARS-CoV-2, el cual es casi imposible que surgiera a partir de una manipulación de laboratorio partiendo de un coronavirus relacionado. Esto se debe a que el dominio de unión al receptor (RBD, por sus siglas en inglés Receptor Binding Domain) en la proteína espiga (S) del SARS-CoV-2, está optimizado para enlazarse a la enzima convertidora de la angiotensina 2 humana de forma distinta a otros coronavirus. Por lo tanto, solo existen dos posibles teorías que pueden explicar el origen del SARS-CoV-2:
- Selección natural en un animal hospedero antes de una trasferencia zoonótica al humano.
- Selección natural en humanos después de la transferencia zoonótica[39].
A la fecha, no se ha identificado un coronavirus hospedado en un animal que sea lo suficientemente similar como para afirmar de manera inequívoca que se trata del precursor directo del SARS-CoV-2. Sin embargo, en los coronavirus pueden presentarse mutaciones, inserciones y deleciones cerca de la unión del complejo proteínico S1-S2. Esto puede ser una evidencia de un proceso evolutivo natural[40]. Para que un coronavirus hospedado en un animal adquiera el sitio de escisión a partir de mutaciones en la proteína S, con la finalidad de unirse a la enzima convertidora de la angiotensina 2 humana, es indispensable que el hospedero animal se encuentre en un ambiente a una densidad de población alta. Esto, con la finalidad de promover una selección natural eficiente y que el coronavirus exprese un gen que codifique a la enzima convertidora de la angiotensina 2 similar a la ortóloga humana.
Por otra parte, de acuerdo con la segunda teoría, también es posible que un precursor del SARS-CoV-2 haya dado un salto zoonótico a los humanos, adquiriendo las características genómicas descritas con anterioridad a través de la adaptación, durante la transmisión no detectada de humano a humano. Una vez adquiridas estas adaptaciones, cabe la posibilidad de que se promoviera la propagación y posteriormente se generara un grupo de casos lo suficientemente grande como para activar el sistema de vigilancia que lo detectó[41].
Los genomas reportados del SARS-CoV-2 indican que todas las cepas aisladas se derivan de un ancestro en común. Adicionalmente, la presencia de un dominio de unión al receptor muy similar a la del SARS-CoV-2 en pangolines hace suponer que probablemente el virus saltó de estos a los humanos. El SARS-CoV-2 tiene una similitud del 91,02% con el β-coronavirus de pangolín malayo a nivel del genoma total, y con una similitud de secuencia de aminoácidos del 97,4% en el dominio de unión al receptor de la proteína S1[42]. El coronavirus de pangolín malayo (M. javanica) y el SARS-CoV-2 tienen cinco residuos de aminoácidos idénticos que son clave en la interacción del coronavirus con el receptor del hospedero. Zhang y colaboradores (2020) llegaron a las mismas conclusiones, señalando que cinco residuos de aminoácidos clave de la proteína S1 de coronavirus del pangolín malayo involucrados en la interacción con la enzima convertidora de la angiotensina 2 humana, son completamente consistentes con los del SARS-CoV-2[43]. Estos resultados permiten tener una certeza alta de que el pangolín malayo es el reservorio del SARS-CoV-2.
Sumado a todo lo anterior, los coronavirus evolucionan y pueden llegar a presentar recombinaciones genéticas ya sea dentro de la especie hospedera o al saltar de una especie a otra. Estas recombinaciones entre distintas cepas de coronavirus pueden conducir a coronavirus mutados con un alto grado de patogenicidad y ser potencialmente más mortales para los humanos[44]. La tasa de mutación en el genoma del SARS-CoV se estimó en 0,80 a 2,38 por 10-3 sustituciones de nucleótidos por sitio, por año[45]. En el SARS-CoV-2, dos mutaciones de la proteína S y la proteína N pueden explicar su transmisión zoonótica[46].
En general, el análisis comparativo de las secuencias genómicas virales es de gran ayuda para rastrear el origen de los coronavirus que infectan al humano y para determinar la relación que guardan con otros virus análogos[47]. A partir de ese tipo de estudios se ha comprobado que casi todos los virus de los grupos a-coronavirus y β-coronavirus se hospedan en mamíferos, y los coronavirus humanos se localizan en alguno de estos grupos, Tabla 2. Los γ-coronavirus, por el contrario, se han aislado únicamente de hospederos aviares. Los δ-coronavirus se han aislado de varias especies de pájaros cantores y de gatos leopardo en China, así como de cerdos[48].
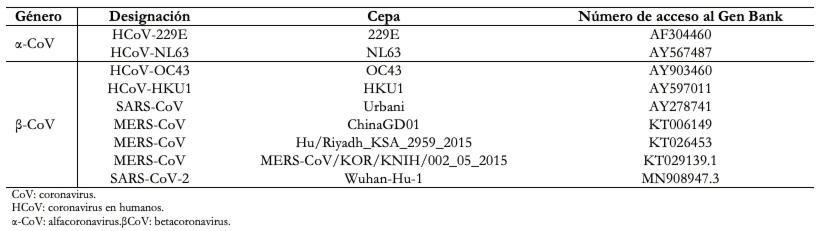 Tamaño completo
Tamaño completo SARS-CoV-2 convertido en una pandemia
Los virus de ácido ribonucleico, como el SARS-CoV-2, tienden a favorecer las pandemias debido a la plasticidad evolutiva inherente a los genomas de ácido ribonucleico, y a la irrupción y alteración que los humanos inducen en los ecosistemas donde ocurren de forma natural los virus. También, los virus de ácido ribonucleico tienen tiempos de generación cortos y tasas evolutivas rápidas[49]. Las tasas evolutivas rápidas de los virus de ácido ribonucleico se desarrollan a partir de ciclos de replicación frecuentes. Esta replicación es susceptible a errores y puede producir poblaciones extensas de genotipos estrechamente relacionados entre sí[50]. Adicionalmente, sin un tratamiento comercial disponible para contener el brote viral y sin una vacuna, estamos obligados a realizar esfuerzos de contención, al menos durante las primeras etapas[51].
El SARS-CoV-2 es el responsable de la denominada enfermedad COVID-19. La aparición de COVID-19 se atribuye a la venta de mariscos y animales salvajes vivos en el mercado de Wuhan, China; que debido a las condiciones imperantes en el sitio, provocó un evento zoonótico.
La propagación del SARS-CoV-2 a nivel mundial se puede explicar con el número reproductivo básico (R0) del virus que es de alrededor de 2,5[52]. Este valor indica que una persona infectará a 2,5 personas en ausencia de medidas preventivas. Para que se produzca una epidemia, el valor de R0 debe ser mayor que la unidad. El valor de R0 para el SARS-CoV-2 en China en las primeras etapas de la epidemia fue de 2,5. A partir de este valor, Anderson y colaboradores (2020) calcularon que en ausencia de medidas de mitigación, aproximadamente el 60% de la población mundial se infectaría[52]. Este es el escenario más catastrófico, sin embargo, en términos reales existen poblaciones menos expuestas que otras. Además, en la mayoría de los países afectados por la pandemia, se han implementado medidas preventivas del contagio. Esto permitirá reducir significativamente la propagación del SARS-CoV-2. También es importante mencionar que conforme avance la pandemia, el factor de reproducción efectiva (Re) tenderá a disminuir hasta llegar a valores por debajo de la unidad, el cual será un indicativo del control de la pandemia. El valor de Re es fuertemente dependiente de las medidas de control. Datos reportados por Kissler y colaboradores (2020) sugieren que el Re ha disminuido hasta valores de alrededor de 1,4[53]. A pesar de ser esto un resultado alentador, es importante señalar que las medidas de distanciamiento social pueden requerir varios meses para lograr controlar de manera efectiva la transmisión y reducir la posibilidad de un rebrote del SARS-CoV-2.
Para entender el impacto de tales medidas, Koo, Cook y Park (2020) desarrollaron un modelo para proyectar el efecto potencial de la intervención de la dispersión del SARS-CoV-2 en Singapur[54]. Estos resultados mostraron que, partiendo de un R0 de 2,5 y considerando una intervención combinada basada en una cuarentena, cierre de escuelas y distanciamiento en el lugar de trabajo; se puede llegar a alcanzar una efectividad del 78,2% para reducir la propagación del virus. Además, los resultados obtenidos por China para contener la propagación del SARS-CoV-2 confirman que las cuarentenas, el distanciamiento social y el aislamiento de las poblaciones infectadas pueden contener la pandemia de manera efectiva[55]. Estas medidas, adicionalmente pueden ayudar a reducir el contagio a grupos de personas que sean altamente susceptibles al SARS-CoV-2. Respecto a esto, Chen y colaboradores (2020) realizaron un estudio donde se hace evidente que personas de edad avanzada y/o con comorbilidades crónicas, tales como enfermedades cerebrovasculares, cardiovasculares, del sistema endócrino, digestivo, respiratorio, nervioso y tumores malignos, pueden llegar a ser más susceptibles de contagiarse y manifestar el estado grave de COVID-19[29].
Personas de todas las edades son susceptibles de infectarse con el SARS-CoV-2. El virus se transmite a través de gotas generadas durante la tos y los estornudos por pacientes sintomáticos y asintomáticos[56]. Los estudios han demostrado mayores cargas virales en la cavidad nasal que en la garganta, sin diferencias en la carga viral entre las personas sintomáticas y asintomáticas[57]. Los pacientes pueden ser infecciosos mientras duren los síntomas e incluso en la recuperación clínica. Las gotas producidas por la tos y los estornudos pueden llegar a alcanzar distancias de uno a dos metros y depositarse en las superficies. El SARS-CoV-2 puede permanecer viable en las superficies durante días en condiciones atmosféricas favorables, pero se destruye rápidamente con desinfectantes comunes como el hipoclorito de sodio y el peróxido de hidrógeno, entre otros[58]. La infección se adquiere, ya sea por inhalación de estas gotas o exponiéndose a superficies contaminadas con el virus. El virus también está presente en las heces y se especula la contaminación del suministro de agua, la cual puede ser una fuente de transmisión[59].
Aun cuando la mayoría de los países en el mundo han tomado medidas de distanciamiento social, los datos publicados el 16 de julio de 2020 por el Coronavirus Resource Center at University of Medicine Johns Hopkins relacionados con COVID-19 reportan 13 758 533 personas infectadas y 589 093 muertos en el mundo. Esto arroja una tasa de letalidad del 4,3%. Algunos países europeos, como Gran Bretaña presentan una tasa de letalidad de 15,4%, alta respecto a la media global; mientras que otros países de Europa presentan una tasa relativamente baja, tal como Alemania con una letalidad del 4,5%. En América, Estados Unidos tiene el mayor número de muertos en todo el mundo, pero con una tasa de letalidad de 3,9%. En América Latina, Brasil tiene el mayor número de infectados (2 012 151) y muertos (76 688), con una tasa de letalidad de 3,8%. México tiene 324 041 infectados y 37 574 muertos, con una letalidad del 11,6%. En América Latina, después de México, el país con la letalidad más alta es Ecuador, 7,3%. Finalmente, Chile el tercer país en América Latina con el mayor número de infectados, reporta 323 698 casos; sin embargo, la letalidad en este país es baja con un 2,2% comparada con la media global. Es importante señalar que la tasa de letalidad permite evaluar el comportamiento de la enfermedad en un punto determinado de la pandemia y varía con respecto al tiempo. La tasa de letalidad reportada aquí se calculó con los datos disponibles al 16 de julio de 2020.
Características y síntomas de COVID-19
La infección por COVID-19 se puede presentar en tres posibles estados: leve, moderado y severo. Los pacientes de menos de 60 años y sin comorbilidades relevantes pueden presentar la enfermedad en un estado leve o moderado. Mientras que los pacientes con comorbilidades significativas pueden presentar un estado moderado. Los casos moderados, además presentan los síntomas de neumonía[60]. Entre los criterios de severidad en adultos estarían, taquipnea (igual o superior a 30 respiraciones en menos de un minuto), menos de 93% de saturación de oxígeno en reposo, el índice PAFI definido como la relación de la presión parcial de oxígeno arterial (PaO2)/fracción de oxígeno inspirado (FiO2)-1 inferior o igual a 300 milímetros de mercurio (si se encuentra por sobre los 1000 metros sobre el nivel del mar, se debe corregir la fórmula)[60].
La OMS recomienda recolectar muestras tanto del tracto respiratorio superior (muestras naso y orofaríngeas) como del tracto respiratorio inferior, esputo expectorado, aspirado endotraqueal o lavado broncoalveolar (BAL, por sus siglas en inglés Bronchoalveolar Lavage). La recolección de muestras de lavado broncoalveolar sólo debe realizarse en pacientes con ventilación mecánica, ya que las muestras del tracto respiratorio inferior parecen seguir siendo positivas durante un período más prolongado. Las muestras requieren almacenamiento a 4 grados Celsius. En esta etapa de identificación de pacientes con COVID-19 se vuelve importante utilizar pruebas de laboratorio confiables para el diagnóstico de la enfermedad. Estas pruebas suelen consistir en ensayos de reacción en cadena de la polimerasa de transcripción inversa en tiempo real o de amplificación isotérmica de ácido nucleico, las cuales se describirán posteriormente.
Las personas con COVID-19 muestran manifestaciones clínicas que incluyen fiebre, fatiga, tos seca, dificultad para respirar y síndrome de dificultad respiratoria aguda. El recuento de glóbulos blancos suele ser normal o bajo. Puede haber linfopenia; un recuento de linfocitos inferior a 1000 se ha asociado con un estado severo. El recuento de plaquetas suele ser normal o levemente bajo. La proteína C reactiva y la velocidad de sedimentación globular generalmente están elevadas, pero los niveles de procalcitonina suelen ser normales. Un nivel alto de procalcitonina puede indicar una coinfección bacteriana. La alanina aminotrasferasa, la aspartato-aminotransferasa, el tiempo de protrombina, la creatinina, el dímero D, la creatina fosfoquinasa y la lactato-deshidrogenasa, pueden estar elevados y los niveles altos están asociados con un estado severo de la enfermedad. En la Tabla 3 se enlistan algunos parámetros de laboratorio clínico de 21 pacientes estudiados en China clasificados como moderados (10 casos) y severos (11 casos) de acuerdo a la Comisión Nacional de China[61]. Cabe señalar que estos síntomas, así como los resultados de laboratorio clínico, no son del todo determinantes para tener una claridad en la gravedad de los enfermos de COVID-19.
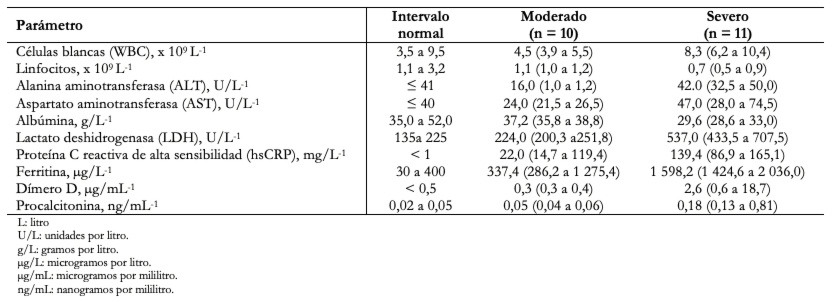 Tamaño completo
Tamaño completo De manera complementaria a los parámetros sanguíneos descritos en la Tabla 3, los pacientes sospechosos de presentar COVID-19 deben someterse a una revisión clínica que incluya la toma de signos vitales, una exploración torácica de rutina y medición de la saturación de oxígeno mediante un oxímetro de pulso. Cabe señalar que COVID-19 no se puede diferenciar de infecciones virales respiratorias, tales como influenza, parainfluenza, virus sincicial respiratorio, entre otros, clínicamente o mediante pruebas de laboratorio rutinarias. Adicionalmente, los pacientes con COVID-19 en estado moderado o severo, idealmente, deben someterse a una tomografía computarizada de alta resolución de tórax para evaluar el grado de afectación. La combinación de las imágenes radiológicas de la tomografía computarizada de tórax con las manifestaciones clínicas y la comprobación anatomopatológica, permiten confirmar la patología pulmonar de COVID-19. La evaluación de una tomografía computarizada de tórax de un paciente con COVID-19 debe contemplar las siguientes características[62]:
- Presencia de opacidades del tipo “vidrio esmerilado”.
- Presencia de consolidación.
- Lateralidad de las opacidades y consolidación en vidrio esmerilado.
- Número de lóbulos afectados donde estaban presentes opacidades de vidrio esmerilado o consolidadas.
- Grado de afectación de cada lóbulo pulmonar además del grado general de afectación pulmonar medido por medio de un "puntaje de gravedad total"[63].
- Presencia de nódulos.
- Presencia de derrame pleural.
- Presencia de linfadenopatía torácica (a partir de un tamaño de ganglio linfático superior a 10 milímetros en dimensión de eje corto).
- Anomalías de las vías respiratorias.
- Distribución axial de la enfermedad.
- Presencia de enfermedad pulmonar subyacente, como enfisema o fibrosis.
Los hallazgos típicos de las imágenes de tomografía computarizada torácica de pacientes con COVID-19 son opacidades bilaterales multifocales del tipo “vidrio esmerilado” irregulares o consolidación con engrosamiento interlobular septal y vascular, principalmente en los campos periféricos de los pulmones. La opacidad de vidrio esmerilado es un área nebulosa de aumento de la atenuación del pulmón con marcas bronquiales y vasculares preservadas. En pacientes con COVID-19, la tasa de incidencia de las opacidades vidrio esmerilado y la consolidación son de aproximadamente 86 y 29%, respectivamente[64]. La morfología más común de las opacidades vidrio esmerilados son las irregulares y redondas, seguidas de las triangulares y lineales[65]. Wu y colaboradores (2020) mencionan que las opacidades de vidrio esmerilado triangulares bajo la pleura con engrosamiento septal interlobular, llamado "telaraña", se consideran otro indicio de COVID-19.
Los hallazgos de la tomografía computarizada de pacientes con COVID-19 pueden cambiar a medida que la enfermedad avanza. En la etapa inicial, la tomografía computarizada torácica muestra pequeñas opacidades de vidrio esmerilado lobulares y subsegmentarias irregulares, cambios intersticiales y engrosamiento de lúmenes vasculares en todo momento. En una etapa posterior y máxima, las lesiones progresan gradualmente a múltiples opacidades de vidrio esmerilado en los pulmones y algunos pacientes pueden tener una consolidación densa en los lóbulos. El tabique interlobular en las opacidades de vidrio esmerilado se engrosa, similar a la señal de "empedrado". En casos severos, los pacientes pueden tener lesiones difusas en ambos pulmones, algunos de los cuales muestran una apariencia "pulmón blanco". Los broncogramas aéreos y los signos de penetración de vasos sanguíneos son comunes. En la etapa de absorción o remisión, las opacidades de vidrio esmerilado y la consolidación se resuelven sin signos de "empedrado", y se pueden llegar a observar lesiones de fibrosis subpleural parenquimatosa. Por lo tanto, la tomografía computarizada puede indicar la evolución de la enfermedad y evaluar la gravedad clínica y el alcance de COVID-19.
Cabe señalar que, en algunas ocasiones, el uso rutinario de tomografía computarizada puede conducir a falsos positivos, por ejemplo, casos de neumonía que no son ocasionados por SARS-CoV-2. Sin embargo, con el transcurso del tiempo es posible que mejore la capacidad predictiva de esta técnica de diagnóstico. También, los pacientes se exponen a un riesgo potencial debido a la radiación empleada durante la adquisición de las imágenes. Otro riesgo importante se debe a una infección cruzada durante el proceso de escaneo. Por tal motivo, es importante tener estrictas medidas de protección durante el escaneo. A pesar de todos estos riesgos, la tomografía computarizada sigue siendo un método importante para el diagnóstico, evaluación del alcance, severidad y seguimiento de COVID-19[66].
Lauer y colaboradores (2020) estimaron la duración del período de incubación de COVID-19. El estudio se realizó a partir de un análisis agrupado de casos confirmados de COVID-19 entre el 4 de enero y el 24 de febrero de 2020 a partir de reportes y comunicados de prensa de 50 provincias, regiones y países, excluyendo a la ciudad de Wuhan, China[67]. En el estudio se tomaron en cuenta las características demográficas del paciente, fechas y horas de posible exposición, inicio de síntomas, inicio de fiebre y hospitalización. Los resultados mostraron 181 casos confirmados con exposición identificable e identificación del inicio de los síntomas para estimar el período de incubación de COVID-19. Con esos resultados se determinó una mediana del período de incubación de cinco días (dentro de un intervalo de confianza del 95%; 4,5 a 5,8 días), y el 97,5% de los infectados que desarrollaron síntomas lo hicieron dentro de los 11,5 días después de haber sido infectados (intervalo de confianza: 8,2 a 15,6 días). Adicionalmente, el estudio permitió estimar que 101 de cada 10 000 casos desarrollarán síntomas después de 14 días de monitoreo o cuarentena de incubación para COVID-19.
Métodos para la detección del SARS-CoV-2
La secuenciación del genoma total del SARS-CoV-2 es uno de los enfoques más completos para la identificación y cuantificación de los ácidos nucleicos virales, con gran exactitud y fiabilidad. Esto permite identificar inequívocamente al SARS-CoV-2. La primera secuencia completa del genoma del SARS-CoV-2 disponible en el GenBank tiene el número de acceso MN908947. Esta secuenciación permitió identificar cinco marcos de lectura abiertos (ORF) típicos en el genoma del SARS-CoV-2, la poliproteína ORF1ab (7 096 aminoácidos), la proteína S (1 273 aminoácidos), la proteína E (75 aminoácidos), la proteína M (222 aminoácidos) y la proteína N (419 aminoácidos). Posteriormente, se han reportado secuencias de otros genes del SARS-CoV-2 empleando las plataformas Illumina y Nanoporo[68]. Debido a que la secuenciación del genoma completo es relativamente costosa, requiere de tiempos prolongados de pruebas y es complicada, no es adecuada para pruebas urgentes y a gran escala. Por tal motivo, se requieren de métodos alternativos que permitan obtener resultados de manera rápida, relativamente barata y confiable.
La reacción en cadena de la polimerasa de transcripción inversa en tiempo real es el método de prueba más popular para la detección de SARS-CoV-2, que comúnmente emplea muestras de la parte posterior de la nariz o la boca del paciente. A partir de estas muestras se extrae el ácido ribonucleico y se convierte en su ácido desoxirribonucleico complementario vía una reacción de transcripción en presencia de la enzima transcriptasa inversa. Posteriormente, los fragmentos de genes específicos dentro del ácido desoxirribonucleico complementario se amplifican usando iniciadores para objetivos específicos. Los iniciadores generalmente se obtienen de tres regiones del genoma viral con secuencias conservadas:
- El gen RdRP (ácido ribonucleico dependiente del gen que expresa al ácido ribonucleico polimerasa) en el marco de lectura abierta de la región ORF1ab.
- El gen que expresa la proteína E.
- El gen que expresa la proteína N.
El ensayo puede diseñarse como un sistema de dos objetivos, donde un iniciador detecta universalmente numerosos coronavirus, incluido el SARS-CoV-2, y un segundo conjunto de iniciadores que solo detecta el SARS-CoV-2[69].
Escutenaire y colaboradores (2007) desarrollaron un ensayo de reacción en cadena de la polimerasa de transcripción inversa en tiempo real para la detección genérica de coronavirus empleando como iniciador la poliproteína ORF1b y el fluoróforo SYBR-Green 1 para la detección[70]. En el estudio se evaluaron los coronavirus humanos HCoV-NL63, HCoV-OC43, HCoV-229E y SARS-CoV. Los resultados mostraron que el ensayo es útil cuando se tiene una gama amplia de virus genéticamente distantes.
Liu y colaboradores (2020) realizaron pruebas de detección de SARS-CoV-2 en 4 880 pacientes de un hospital de Wuhan, China, empleando una técnica de reacción en cadena de la polimerasa de transcripción inversa en tiempo real. Los resultados del estudio mostraron que al utilizar muestras de líquido de lavado broncoalveolar y usar como objetivos los genes fragmento de la poliproteína ORF1ab, se logra una tasa de detección del 100% del SARS-CoV-2.
Kasteren y colaboradores (2020) compararon el rendimiento analítico y clínico básico de kits de reacción en cadena de la polimerasa de transcripción inversa en tiempo real seleccionados de siete fabricantes diferentes (Altona Diagnostics, BGI, CerTest Biotec, KH Medical, Primer Design, R Biopharm AG y Seegene)[71]. Los kits se pueden localizar en el siguiente sitio web (accesado el 22 de julio de 2020). Los resultados del estudio mostraron una eficacia de la reacción en cadena de la polimerasa superiores al 96% para todos los ensayos y un límite de detección de 98% que varió dentro de un intervalo de seis veces para los diversos objetivos de los kits de reacción en cadena de la polimerasa de transcripción inversa en tiempo real.
Por otra parte, los métodos de amplificación isotérmica más conocidos son la amplificación mediada por transcripción o replicación de secuencia autosostenida, amplificación basada en secuencia de ácido nucleico, amplificación mediada por señal de tecnología de ácido ribonucleico, amplificación desplazamiento de cadena, amplificación de círculo rodante, amplificación isotérmica de ácido desoxirribonucleico mediada por bucle, amplificación de desplazamiento múltiple isotérmico, amplificación dependiente de helicasa, amplificación isotérmica de iniciador único y amplificación dependiente de helicasa circular[72]. Una técnica de amplificación isotérmica de ácido nucleico más recientemente empleada está basada en las repeticiones palindrómicas cortas agrupadas regularmente entre espacios intercalados, comúnmente conocida como CRISPR. Estas secuencias pueden ser reconocidas y cortadas por un conjunto de enzimas bacterianas, llamadas enzimas asociadas a repeticiones palindrómicas cortas agrupadas y regularmente espaciadas, ejemplificadas por las familias de enzimas Cas12 y Cas13. Estas dos familias de enzimas pueden programarse para atacar y cortar secuencias de ácidos ribonucleicos virales[73].
Lin y colaboradores (2020) desarrollaron un ensayo basado en la amplificación isotérmica mediada por bucle (amplificación isotérmica de ácido desoxirribonucleico mediada por bucle) combinada con la transcripción inversa (amplificación isotérmica de ácido desoxirribonucleico mediada por bucle en tiempo real) que permite la detección directa de ácido ribonucleico de SARS-CoV-2[74]. Los autores llamaron al método iso-thermal LAMP based method for COVID-19 (iLACO). Este método mostró un desempeño similar con muestras de ácido ribonucleico o ácido desoxirribonucleico complementario de SARS-CoV-2. El ensayo iso-thermal LAMP based method for COVID-19 es de costo bajo, rápido, robusto, simple y preciso para la detección de SARS-CoV-2.
Broughton y colaboradores (2020) desarrollaron y realizaron la validación inicial de un ensayo de repeticiones palindrómicas cortas agrupadas y regularmente espaciadas basado en la enzima Cas12 para la detección del SARS-CoV-2 a partir de muestras de ácido ribonucleico[75]. El ensayo se denominó transformador de repeticiones palindrómicas cortas agrupadas y regularmente espaciadas (CRISPR) dirigido por endonucleasa de ácido desoxirribonucleico (ADN) de SARS-CoV-2, o DETECTR. Este ensayo realiza la transcripción inversa y la amplificación isotérmica simultáneamente utilizando una amplificación mediada por bucle (amplificación isotérmica de ácido desoxirribonucleico mediada por bucle en tiempo real)[76] para ácido ribonucleico nasofaríngeo u orofaríngeo, seguido de la detección por Cas12 de secuencias predefinidas de coronavirus, después de lo cual la escisión de una molécula reportera confirma la detección del virus. El ensayo DETECTR basado en repeticiones palindrómicas cortas agrupadas y regularmente espaciadas es cualitativo y sólo requiere de alrededor de 45 minutos, con predicción positiva del 95% y una predicción negativa del 100%.
Otro tipo de pruebas son las serológicas que se caracterizan por el análisis de sangre, suero o plasma sanguíneo, saliva, esputo y otros fluidos donde se encuentren las moléculas objetivo. Estas pruebas tienen como fundamento reacciones antígeno-anticuerpo que provocan cambios de color mediante el empleo de un conjugado enlazado a una enzima y un sustrato enzimático que sirve para identificar la presencia y concentración de proteínas[77]. Estos ensayos se denominan inmunoensayos enzimáticos o ensayos de inmunosorbentes de enzimas enlazadas (Enzyme-Linked Immuno Sorbent Assay, ELISA).
Los anticuerpos generados por el hospedero en respuesta a las proteínas virales pueden estar presentes durante un período de tiempo prolongado y esto permite detectar indirectamente el SARS-CoV-2 de manera confiable. Las pruebas de anticuerpos pueden ser particularmente útiles para dar seguimiento al COVID-19. Las técnicas serológicas basadas en anticuerpos pueden ser pruebas cualitativas o cuantitativas. Las pruebas cualitativas más comúnmente empleadas consisten en la detección de las inmunoglobulinas A, Inmunoglobulina G e Inmunoglobulinas.
Guo y colaboradores (2020) desarrollaron un método para el diagnóstico de COVID-19 que consistió en evaluar la respuesta humoral contra el SARS-CoV-2, incluida la respuesta de Inmunoglobulinas A, Inmunoglobulinas M e Inmunoglobulinas G, empleando un ensayo basado en inmunosorbentes de enzimas enlazadas de la proteína nucleocápside viral recombinante[78]. El estudio involucró una muestra de líquido de lavado broncoalveolar (BALF, por sus siglas en inglés Bronchoalveolar Lavage Fluid). Los resultados de este estudio demostraron que el plasma de pacientes con SARS-CoV-2 no tienen reactividad cruzada con otros coronavirus, excepto con el SARS-CoV. La prueba con Inmunoglobulinas mostró una efectividad alta para la detección de SARS-CoV-2 después de 14 (rango intercuartílico, de 10 a 18) días de la aparición de los síntomas.
Infantino y colaboradores (2020) emplearon un inmunoensayo quimioluminiscente específico para los anticuerpos Inmunoglobulinas M e Inmunoglobulinas G anti-SARS-CoV-2, denominado ensayo de inmunoabsorción ligado a enzimas e inmunoensayo de quimioluminiscencia[79]. Los resultados del estudio mostraron que la mayoría de los pacientes con COVID-19 estudiados tienen tanto anticuerpos Inmunoglobulinas M como Inmunoglobulinas G. La sensibilidad total del ensayo fue de 75%. Los anticuerpos Inmunoglobulina G mostraron una especificidad del 100%. Esto hace que el ensayo sea apropiado para diferentes protocolos de evaluación de inmunidad de los pacientes.
En la Tabla 4 se muestran los métodos de diagnóstico de COVID-19 más representativos y algunas de sus características.
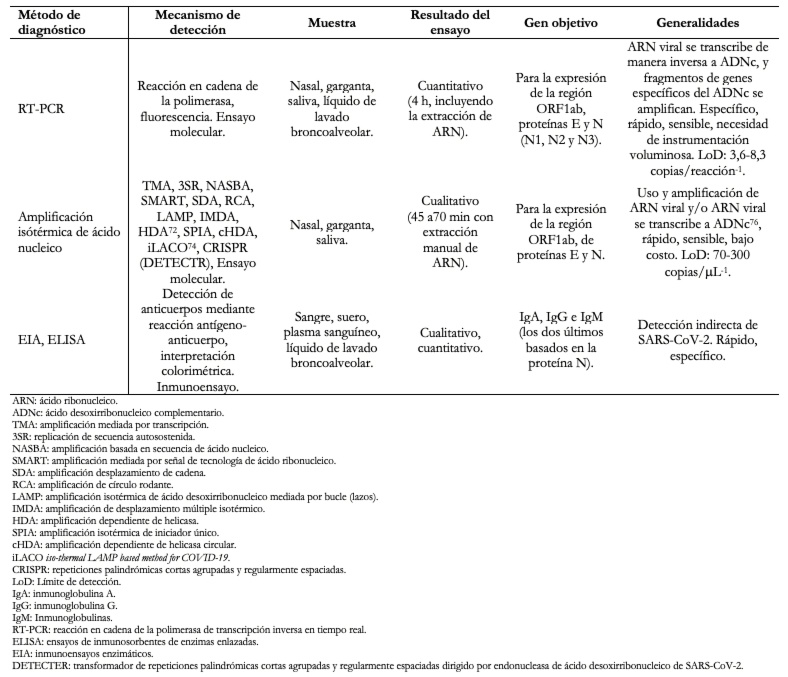 Tamaño completo
Tamaño completo La detección y monitoreo del SARS-CoV-2 se debe realizar empleando dos pruebas, en distintas etapas de la infección, para asegurar que las personas que fueron infectadas con el virus ya no son portadoras de este. La primera prueba se debe emplear para detectar personas que fueron infectadas con el SARS-CoV-2 empleando pruebas serológicas[78],[79]. La segunda prueba debe utilizarse para detectar el material genético viral del SARS-CoV-2 en el paciente mediante una reacción en cadena de la polimerasa de transcripción inversa en tiempo real[70],[71] o una amplificación isotérmica de ácido nucleico[74],[75].
Antivirales evaluados para combatir el SARS-CoV-2
A pesar del esfuerzo global realizado para desarrollar medicamentos o vacunas para tratar o evitar la infección con el SARS-CoV-2, actualmente no existe un antiviral comprobado para controlar el brote de COVID-19 o inactivar al coronavirus. Por lo que los fármacos usados no son del todo efectivos o los estudios publicados no dan certeza de su efectividad. La estrategia actual para tratar a pacientes con COVID-19 incluyen además prevención de infecciones, medidas clínicas de apoyo para pacientes severos con oxígeno y soporte con ventilador mecánico[80]. Wang y colaboradores (2020) realizaron pruebas antivirales contra el SARS-CoV-2 empleando células Vero E6 que infectaron con SARS-CoV-2 a una multiplicidad de infección de 0,05 en el tratamiento con diferentes dosis de remdesivir y cloroquina[81]. Las células Vero E6 son células epiteliales de riñón de un mono verde africano que pertenecen a un linaje celular continuo y aneuploidico. Esto significa que las células Vero E6 se replican a través de muchos ciclos de replicación sin envejecer con un número anormal de cromosomas y se emplean para la producción de vacunas contra enfermedades virales. Mientras que el remdesivir es un pro-fármaco antiviral que pertenece al grupo de análogos de nucleótidos, que se incorpora a las cadenas de ácido ribonucleico virales nacientes.
Los resultados de este estudio mostraron que el valor de concentración efectiva al 90% del remdesivir contra SARS-CoV-2 en las células Vero E6 fue de 1,76 micrometros. Por otra parte, en ese estudio también se evaluó la actividad antiviral frente al SARS-CoV-2 de la cloroquina, este último es un fármaco antipalúdico ampliamente utilizado. La cloroquina bloquea la infección por el virus al aumentar el pH endosómico requerido para la fusión virus/célula. El estudio demostró que la cloroquina funcionaba tanto en el contagio como en las etapas posteriores. Además de su actividad antiviral, la cloroquina tiene una actividad inmunomoduladora, que puede aumentar sinérgicamente su efecto antiviral in vivo. La cloroquina se distribuye ampliamente en todo el cuerpo, incluidos los pulmones, después de la administración oral. El valor de concentración efectiva al 90% de la cloroquina contra el SARS-CoV-2 en células Vero E6 fue de 6,90 micrometros.
Cabe señalar que la Food and Drug Administration recientemente autorizó el uso de emergencia del remdesivir, el fosfato de cloroquina y el sulfato de hidroxicloroquina para el tratamiento de COVID-19 en pacientes que se encuentren hospitalizados.
Adicionalmente, una prueba francesa a escala pequeña sugiere que la hidroxicloroquina y el antibiótico azitromicina, ayudan a los pacientes con COVID-19 a recuperarse más rápido[82]. Los pacientes recibieron 600 miligramos de hidroxicloroquina diariamente y su carga viral en hisopos nasofaríngeos fue analizada diariamente en un hospital. Dependiendo de su presentación clínica, se agregó azitromicina al tratamiento. Los pacientes no tratados de otro centro y los casos que rechazaron el protocolo se incluyeron como controles negativos. El tratamiento con hidroxicloroquina por sí solo pareció eliminar el virus en 14 de 20 personas que recibieron el medicamento durante seis días, en comparación con solo dos de 16 personas en el grupo de control. Seis personas que recibieron hidroxicloroquina junto con azitromicina se reportaron "curadas virológicamente" en un período de seis días. A pesar del tamaño de muestra pequeño, este estudio sugiere que el tratamiento con hidroxicloroquina se asocia significativamente con la reducción-desaparición de la carga viral en pacientes con COVID-19 y su efecto se ve reforzado por la azitromicina. Un estudio más reciente, realizado por Chen y colaboradores (2020) con 62 pacientes, 31 de ellos tratados con hidroxicloroquina (Tabla 5), mostró que el 80,6% de los pacientes tratados presentaron una mejoría notable[83]. Estos resultados son prometedores. Sin embargo, se requiere la implementación de pruebas clínicas aleatorizadas con un monitoreo apropiado para determinar si la cloroquina y la hidroxicloroquina previenen o tratan de manera eficaz al COVID-19[84].
A pesar de todos los potenciales efectos positivos que se le pueden atribuir a la cloroquina y la hidroxicloroquina, así como su amplia distribución en el mercado a costos bajos; es importante señalar que estos fármacos tienen un margen muy estrecho entre la dosis terapéutica y la de intoxicación. Ambos fármacos se han asociado a trastornos cardiovasculares que pueden poner en riesgo la vida del paciente. Por lo tanto, el uso de la cloroquina y la hidroxicloroquina debe estar sujeto a reglas estrictas y no se recomienda la automedicación[85]. Adicionalmente, es importante señalar que hasta la fecha no se han realizado el número suficiente de estudios aleatorizados a doble ciego, y no se ha establecido la eficacia clínica efectiva de estos fármacos en el tratamiento de la infección por SARS-CoV-2[86].
Otro estudio prometedor para el tratamiento del COVID-19 fue recientemente reportado por Zhou y colaboradores (2020) y consistió en el uso del interferon-a2b con o sin umifenovir. Este tratamiento redujo la duración del virus detectable en el tracto respiratorio superior y, en paralelo, redujo la duración de los niveles sanguíneos elevados de los marcadores inflamatorios interleucina -6 y proteína C reactiva con o sin el umifenovir. El interferon-a2b es un antiviral de amplio espectro que exhibe efectos inhibitorios directos sobre la replicación viral y refuerza una respuesta inmune a la infección por un virus. El mecanismo de antiproliferación del interferon-a2b se produce a través de la regulación de la síntesis de proteínas y la traducción selectiva de proteínas implicadas en la inhibición de la proliferación[87].
En la Tabla 5 se enlistan los estudios de series de casos clínicos con algunos de los fármacos que han involucrado el mayor número de pacientes y han mostrado resultados prometedores. Seguramente estos trabajos conducirán a pruebas con poblaciones de pacientes más grandes en un futuro próximo.
 Tamaño completo
Tamaño completo Por otra parte, Richardson y colaboradores (2020) emplearon BenevolentAI, un enorme depósito de información médica estructurada, que incluye numerosas conexiones extraídas de la literatura científica mediante inteligencia artificial, junto con una búsqueda específica para SARS-CoV-2, con la finalidad de identificar medicamentos antivirales aprobados con la potencial capacidad de bloquear el proceso de infección viral[90]. A partir de ese estudio in silico, Richardson y colaboradores (2020) encontraron al baricitinib, un inhibidor selectivo y reversible de las Janus quinasa JAK1 y JAK2. El baricitinib se emplea en el tratamiento de la artritis reumatoide en adultos. El estudio predice que este fármaco reduce la capacidad del virus para infectar células pulmonares con el SARS-CoV-2.
Finalmente, en relación a la búsqueda de agentes terapéuticos y vacunas para coronavirus humano, Liu y colaboradores (2020) realizaron una revisión profunda de artículos de investigación y patentes[91]. Las patentes revisadas destacan las estrategias antivirales que involucran moléculas pequeñas y productos biológicos dirigidos a interacciones moleculares complejas involucradas en la infección y replicación de los coronavirus. Los productos biológicos incluyen anticuerpos terapéuticos, citocinas y terapias basadas en ácidos nucleicos dirigida a la expresión génica del virus, así como a varios tipos de vacunas. Consecuentemente, Liu y colaboradores (2020) proporcionan las bases para el desarrollo continuo de agentes terapéuticos y vacunas relacionados con los coronavirus humanos.
Con relación a esto, Jackson y colaboradores (2020) reportaron hallazgos preliminares de la evaluación de una vacuna en fase 1 para determinar la seguridad e inmunogenicidad de una vacuna mRNA SARS-CoV-2. La fase 1 involucró a 45 participantes saludables de 18 a 55 años de edad, quienes recibieron la candidata a vacuna en uno de tres niveles de dosis (25, 100 o 250 microgramos) suministradas en dos vacunaciones con 28 días de diferencia[92]. Los resultados del estudio mostraron que después de la primera vacunación, la concentración de anticuerpos, medidos el día 29, fue más alta a dosis más altas. Después de la segunda vacunación, los anticuerpos medidos el día 57 siguieron aumentando con muy buenos resultados en los pacientes que recibieron las dosis de 250 microgramos, incrementando 5,6 veces la cantidad de anticuerpos respecto a la primera vacunación. En el estudio también lograron detectar actividad neutralizadora después de la segunda vacunación. Cabe señalar que en los participantes se presentaron algunos efectos adversos que incluyeron fatiga, escalofríos, dolor de cabeza, mialgia y dolor en el lugar de la inyección después de la segunda vacunación.
Conclusiones
La propagación del SARS-CoV-2 se ha extendido a prácticamente todo el mundo, ocasionando fuertes afectaciones al sector salud. Las vías de transmisión del virus son principalmente de humano a humano. Sin embargo, existen algunos estudios recientes que señalan su posible transmisión a través del aire y el agua. El grupo de personas de mayor riesgo por exposición al virus son los adultos mayores a 60 años. Personas de este grupo con alguna enfermedad crónica son altamente susceptibles de presentar el estado severo de COVID-19 y requieren de hospitalización.
El diagnóstico de SARS-CoV-2 puede estar acompañado de imágenes de tomografía computarizada de tórax para evaluar el grado de afectación del paciente. Por otra parte, la mitigación significativa de la propagación del virus se puede lograr con la implementación de una intervención combinada basada en una cuarentena, cierre de escuelas y distanciamiento en el lugar de trabajo. Estas medidas, de acuerdo a modelados de la propagación del virus, pueden lograr que la dispersión se reduzca en aproximadamente un 78%. Esto depende, además de las medidas anteriores, de la disponibilidad de pruebas rápidas, confiables y baratas para monitorear a los pacientes infectados y aquellos altamente susceptibles de contagio.
Para la detección del SARS-CoV-2 se recomiendan las pruebas reacción en cadena de la polimerasa de transcripción inversa en tiempo real, iso-thermal LAMP based method for COVID-19 y DETECTR.
Respecto a los tratamientos de COVID-19, a pesar de existir algunos casos de estudio con cloroquina, hidroxicloroquina, remdesivir, interferon-a2b y oseltamivir en pacientes hospitalizados, aún es imposible afirmar que se dispone de tratamientos que combatan de manera eficiente y confiable al SARS-CoV-2. En la actualidad se están realizando estudios clínicos aleatorizados con los fármacos antes mencionados y próximamente se conocerá su impacto real.
Finalmente, cabe mencionar que el conocimiento que se está generando y su libre disposición desde prácticamente todas las editoriales del mundo, en torno al SARS-CoV-2 está permitiendo avanzar a una velocidad nunca vista en el seguimiento y control de COVID-19, una enfermedad que ha conmocionado al mundo entero.
Corrección
Modificamos la versión en español de este artículo el 18/11/2020 para hacer las siguientes correcciones:
Se modificó el título en inglés:
Origin, structural characteristics, prevention measures, diagnosis and potential drugs to prevent and COVID-19
Por:
Origin, structural characteristics, prevention measures, diagnosis and potential drugs to prevent and control COVID-19
Se modificó el párrafo:
Los anticuerpos generados por el hospedero en respuesta a las proteínas virales pueden estar presentes durante un período de tiempo prolongado y esto permite detectar indirectamente el SARS-CoV-2 de manera confiable. Las pruebas de anticuerpos pueden ser particularmente útiles para dar seguimiento al COVID-19. Las técnicas serológicas basadas en anticuerpos pueden ser pruebas cualitativas o cuantitativas. Las pruebas cualitativas más comúnmente empleadas consisten en la detección de las inmunoglobulinas A, Inmunoglobulina G e Inmunoglobulinas.
Por:
Los anticuerpos generados por el hospedero en respuesta a las proteínas virales pueden estar presentes durante un período de tiempo prolongado y esto permite detectar indirectamente el SARS-CoV-2 de manera confiable. Las pruebas de anticuerpos pueden ser particularmente útiles para dar seguimiento al COVID-19. Las técnicas serológicas basadas en anticuerpos pueden ser pruebas cualitativas o cuantitativas. Las pruebas cualitativas más comúnmente empleadas consisten en la detección de las inmunoglobulina A, Inmunoglobulina G e Inmunoglobulina M.
Se modificó el párrafo:
Los resultados de este estudio mostraron que el valor de concentración efectiva al 90% del remdesivir contra SARS-CoV-2 en las células Vero E6 fue de 1,76 micrometros. Por otra parte, en ese estudio también se evaluó la actividad antiviral frente al SARS-CoV-2 de la cloroquina, este último es un fármaco antipalúdico ampliamente utilizado. La cloroquina bloquea la infección por el virus al aumentar el pH endosómico requerido para la fusión virus/célula. El estudio demostró que la cloroquina funcionaba tanto en el contagio como en las etapas posteriores. Además de su actividad antiviral, la cloroquina tiene una actividad inmunomoduladora, que puede aumentar sinérgicamente su efecto antiviral in vivo. La cloroquina se distribuye ampliamente en todo el cuerpo, incluidos los pulmones, después de la administración oral. El valor de concentración efectiva al 90% de la cloroquina contra el SARS-CoV-2 en células Vero E6 fue de 6,90 micrometros.
Por:
Los resultados de este estudio mostraron que el valor de concentración efectiva al 90% del remdesivir contra SARS-CoV-2 en las células Vero E6 fue de 1,76 micromolar. Por otra parte, en ese estudio también se evaluó la actividad antiviral frente al SARS-CoV-2 de la cloroquina, este último es un fármaco antipalúdico ampliamente utilizado. La cloroquina bloquea la infección por el virus al aumentar el pH endosómico requerido para la fusión virus/célula. El estudio demostró que la cloroquina funcionaba tanto en el contagio como en las etapas posteriores. Además de su actividad antiviral, la cloroquina tiene una actividad inmunomoduladora, que puede aumentar sinérgicamente su efecto antiviral in vivo. La cloroquina se distribuye ampliamente en todo el cuerpo, incluidos los pulmones, después de la administración oral. El valor de concentración efectiva al 90% de la cloroquina contra el SARS-CoV-2 en células Vero E6 fue de 6,90 micromolar.

