Comunicaciones breves
← vista completaPublicado el 3 de octubre de 2023 | http://doi.org/10.5867/medwave.2023.09.2735
Análisis poblacional in silico del haplotipo CLOCK rs3749474T-rs4864548A y su relación con obesidad
In silico population analysis of the CLOCK rs3749474T-rs4864548A haplotype and its relationship with obesity
Resumen
Se ha sugerido que el haplotipo rs3749474T/rs4864548A del gen CLOCK aumentaría el riesgo de obesidad, pero se desconoce el patrón de variabilidad poblacional de estos alelos y del haplotipo. El objetivo de este estudio es determinar el nivel de ligamiento entre los alelos de riesgo rs3749474T y rs4864548A a partir de la base de datos 1000Genomes para confirmar la existencia del haplotipo TA de los polimorfismos rs3749474-rs4864548 del gen CLOCK y su frecuencia cinco macro poblaciones. Se analizó el desequilibrio de ligamiento y las frecuencias haplotípicas para 2504 individuos, de 26 poblaciones, utilizando el estadístico r y la prueba exacta de Fisher. Existe una alta frecuencia del haplotipo TA en Latinoamérica (44,8%), un alto desequilibrio de ligamiento (r= 0,92) a nivel mundial entre esos alelos, una alta diferenciación entre macro poblaciones y una alta homogeneidad al interior de ellas. La evidencia presentada permite sugerir la realización de posteriores estudios de asociación entre este haplotipo y el nivel de riesgo de obesidad y sobrepeso en poblaciones latinoamericanas.
Ideas clave
- El haplotipo rs3749474T/rs4864548A del gen CLOCK aumentaría el riesgo de obesidad, pero se desconoce su patrón de variabilidad poblacional.
- Este estudio aporta antecedentes acerca de la frecuencia del haplotipo rs3749474T/rs4864548A del gen CLOCK en cinco macro poblaciones y 26 poblaciones.
- Existe una alta frecuencia del haplotipo TA en Latinoamérica, un alto desequilibrio de ligamiento, una alta diferenciación entre macro poblaciones y una alta homogeneidad al interior de ellas.
- Este estudio está limitado al análisis in silico de bases de datos, por lo que se sugiere confirmar la asociación del haplotipo TA con obesidad a través de estudios con datos primarios.
Introducción
El gen CLOCK es uno de los genes que, a través de su proteína codificada, regula los ciclos circadianos, cuyos polimorfismos se han asociado al desarrollo de la obesidad, hipercolesterolemia, hiperglicemia entre otras condiciones [1]. Los SNPs rs3749474T y rs4864548A del gen CLOCK se han vinculado en forma independiente a un mayor índice de masa corporal, aumentando el riesgo de sobrepeso u obesidad hasta en 1,5 veces [2,3,4,5,6].
En el estudio realizado por Pino-Astorga et al se encontró el caso de un sujeto que presentaba ambos polimorfismos (rs3749474T y rs4864548A), cuyo perfil antropométrico lo sitúa como de alto riesgo debido a su sobrepeso y circunferencia de cintura, a pesar de tener hábitos alimenticios normales [7]. Lo anterior sugiere un efecto aditivo entre ambos alelos de riesgo, sugiriendo la existencia de del haplotipo TA para ellos.
El objetivo de esta investigación es determinar el nivel de ligamiento entre los alelos de riesgo rs3749474T y rs4864548A a partir de la base de datos 1000genome para confirmar la existencia del haplotipo TA de los polimorfismos rs3749474-rs4864548 del gen CLOCK, como un estudio preliminar a la realización de pruebas de campo confirmatorias en población latinoamericana.
Métodos
Muestra
Se obtuvo los genotipos de los SNPs rs3749474 y rs4864548 del gen CLOCK de 2504 individuos, desde la base de datos del proyecto 1000Genomes en su fase 3 [8]. Para esto, se descargó el total de datos del cromosoma 4, para luego extraer los genotipos rs3749474 y rs4864548 para cada individuo, usando el software VCFtools. En dicho proyecto, todos los genomas fueron secuenciados a baja cobertura (4x)- Para la validación de datos, 24 de ellos fueron secuenciados con alta cobertura (50x). En este estudio se incluyeron las cinco macro poblaciones comprendidas en 1000Genomes: África, Este Asiático, Sur Asiático, Europa y Latinoamérica, junto a las 26 poblaciones dentro de ellas (Tabla 1).
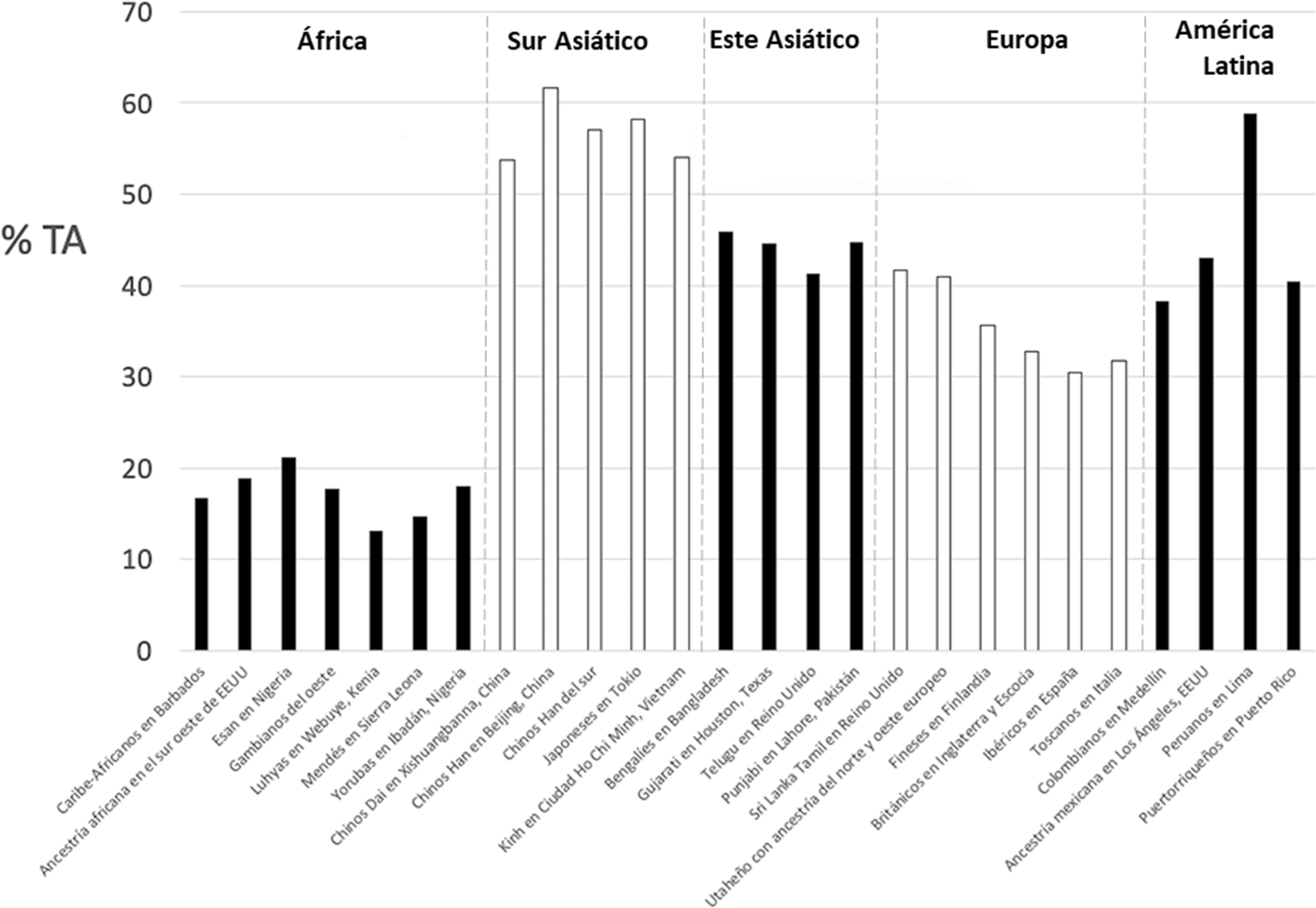
Figura 1: Frecuencia del haplotipo TA en cada una de las poblaciones y macro poblaciones analizadas en este estudio.
El tamaño muestral para un margen error de 5% y un intervalo de confianza de 95% es de n = 385. Solo una macro población, la latinoamericana, presenta un tamaño muestral bajo ese valor, correspondiendo a 347 participantes. Para ese tamaño de muestra, el margen de error es de 5,15%, es decir muy próximo al 5%.
Análisis estadístico
La base 1000Genomes, en la actual fase de desarrollo contiene información “en fase” de los genotipos. Es decir, es posible reconstruir la distribución de alelos en cada cromosoma parental. Así, usando un algoritmo escrito en R, se identificaron los alelos de ambos SNPs para los 5008 cromosomas analizados, para luego estimar la frecuencia de los cuatro haplotipos según poblaciones y macro poblaciones.
Considerando los alelos ancestrales y derivados para ambos SNPs, así como sus respectivas frecuencias alélicas, se determinaron los haplotipos recombinantes y no-recombinantes, así como sus respectivas frecuencias poblacionales. Luego, se estimó la frecuencia de recombinación y el desequilibrio de ligamiento a través del estadístico r2. Las diferencias en las frecuencias de los haplotipos se analizaron usando la prueba exacta de Fisher.
Resultados
Se obtuvo las siguientes frecuencias a nivel mundial, para los cuatro haplotipos de los SNPs rs3749474 y rs4864548: TA = 37,6%; CG = 62,2%; CA = 0,08% y TG = 0,08%. Considerando esas frecuencias, y que el haplotipo CG es ancestral según la información en 1000Genomes, resulta evidente que TA y CG son los haplotipos parentales en las actuales poblaciones. Así, la frecuencia de recombinación para estos dos loci es de 0,16 %. El desequilibrio de ligamiento fue alto (r2 = 0,92).
La frecuencia de TA en las poblaciones se encuentra en la Tabla 1 y Figura 1. La menor frecuencia de genotipos portadores del haplotipo TA se encuentra en África, seguida por Europa, Latinoamérica, Sur Asiático y Este Asiático. Al comparar las frecuencias genotípicas entre todos los pares de poblaciones, se confirma la alta divergencia de África. Existen diferencias significativas (p < 0,05) en todas las comparaciones, excepto entre Latinoamérica y Sur Asiático (p < 0,62). Existe alta homogeneidad dentro de las cinco macro poblaciones. Perú presenta una alta frecuencia, estadísticamente significativa, de TA en relación con las otras poblaciones de Latinoamérica (p < 0,05).
Discusión
El alto nivel de ligamiento entre los alelos de riesgo rs3749474T y rs4864548A del gen CLOCK, aquí reportado, refuerza la observación del estudio de Pino-Astorga et al [7], pues ambos alelos de riesgo se heredan en conjunto en aproximadamente un 44,8% de los cromosomas en la población latinoamericana, mientras que en el restante 55,2% lo hace el haplotipo de no riesgo CG.
La alta divergencia de la macro población de África, respecto a las otras, podría deberse a la interacción de patrones dietarios y adaptación biológica [9], así como a cambios dietarios experimentados durante la transición alimentaria [10]. Las diferencias entre las restantes cuatro macro poblaciones podrían deberse a presiones selectivas específicas, durante el poblamiento de los diferentes continentes.
Llama la atención la alta homogeneidad entre poblaciones dentro de cada macro población, lo que refuerza nuestra hipótesis sobre la heterogeneidad entre macro poblaciones. La alta divergencia observada en la población de Lima, que exhibió una alta frecuencia del haplotipo TA, requiere de posteriores análisis. Una posibilidad de explicación a este hecho es la mayor acumulación de alelos de origen asiático, considerando la historia de poblamiento y mestizaje ocurrida en Perú.
Una limitación del presente trabajo es la existencia de particularidades poblacionales que aquí no son abarcadas. Ejemplo de ello es la existencia de diferentes ancestrías dentro de las macro poblaciones, como es el caso de poblaciones indígenas en Latinoamérica. Esta necesidad se hace evidente al observar la alta frecuencia del haplotipo TA en población de Lima, Perú. Esta presenta una mayor ancestría nativo americana, en relación con las otras poblaciones latinoamericanas aquí estudiadas. No obstante esta limitación, la evidencia recolectada permite establecer la existencia del haplotipo TA rs3749474-rs4864548 con una relativa alta frecuencia y desequilibrio de ligamiento, siendo un marcador candidato para un estudio de riesgo de sobrepeso y obesidad en población latinoamericana.
Conclusiones
El alto nivel de ligamiento entre los alelos de riesgo rs3749474T y rs4864548A, junto a su relativa alta frecuencia en población latinoamericana, permiten confirmar la existencia del haplotipo TA para estos polimorfismos. Ello lo convierte en un candidato de investigación a nivel población, para establecer su relación con el desarrollo de la obesidad y sobrepeso.

