Artículo de revisión
← vista completaPublicado el 24 de abril de 2023 | http://doi.org/10.5867/medwave.2023.03.2619
Efecto de la epigenética en la artritis reumatoide
Effect of epigenetics on rheumatoid arthritis
Resumen
La artritis reumatoide es una enfermedad autoinmune e inflamatoria que afecta de manera predominante a las articulaciones diartrodiales. En esta patología los factores ambientales o conductuales pueden actuar en sinergia con la predisposición genética, acelerando el inicio y la gravedad de la enfermedad. Este vínculo entre el medio ambiente y el genoma está mediado por marcas epigenéticas en el ácido desoxirribonucleico, incluyendo su metilación, la modificación de histonas y la regulación mediada por ácido ribonucleico no codificante. La epigenética puede generar cambios fenotípicos hereditarios, que no están determinados por modificaciones en la secuencia del ácido desoxirribonucleico y, en consecuencia, son reversibles. Por lo tanto la dieta, los medicamentos y otros factores ambientales, tendrían la capacidad de modularlos. La identificación de una desregulación epigenética específica, puede ofrecer una mayor comprensión de la fisiopatología de la enfermedad e influenciar positivamente en la prevención, diagnóstico y desarrollo de nuevas dianas terapéuticas
Ideas clave
- La artritis reumatoide es una condición médica de carácter autoinmune, inflamatorio y crónico, que puede generar discapacidad.
- Los avances terapéuticos y la clinimetría han permitido un mejoramiento en el control de la enfermedad.
- El conocimiento de la epigenética ha generado una serie de elementos, mejorando la conceptualización del modelo fisiopatológico de la artritis reumatoide e integrando la interacción entre gen y ambiente.
- En esta revisión narrativa evaluamos la evidencia sobre los avances de la epigenética y su efecto en la artritis reumatoide.
Introducción
En la patogenia de la artritis reumatoide participan las células del sistema inmunitario innato como los macrófagos, las células dendríticas y las células citotóxicas naturales. Por parte del sistema inmunitario adaptativo encontramos a los linfocitos T y B, que asociadamente intervienen otras células no inmunitarias como los fibroblastos y células endoteliales [1].
Los estudios en familiares de primer grado apoyan la influencia de la herencia entre el 50 y 60%, la heredabilidad no es modificada por el sexo, y la evidencia sugiere que la artritis reumatoide seropositiva con factor reumatoide y/o anticuerpos anti-péptido cíclico citrulinado es más frecuente en pacientes con antecedentes familiares, en comparación con los que presentan artritis reumatoide seronegativa [2].
Los estudios de asociación del genoma han caracterizado más de100 factores genéticos para la artritis reumatoide, siendo el epítopo compartido el factor de riesgo genético más conocido, especialmente en el fenotipo seropositivo [3,4]. Existen otros genes no antígeno del leucocito humano estudiados como la proteína tirosina fosfatasa no receptora tipo 22 (PTPN22) [5,6] representando en este grupo de moléculas la de mayor susceptibilidad para artritis reumatoide, la cual está relacionada con riesgo para otras enfermedades autoinmunes como diabetes mellitus tipo 1, artritis idiopática juvenil, lupus eritematoso sistémico, síndrome de Sjogren, enfermedad tiroidea autoinmune, vitíligo y diferentes formas de vasculitis. Igualmente se ha analizado polimorfismo del gen antígeno-4 de linfocitos T citotóxicos (CTLA-4) [7,8] y la desregulación del receptor de interleucina-23 (IL-23R) como predisposición para artritis reumatoide, y otras condiciones reumáticas como la espondilitis anquilosante y lupus eritematoso sistémico [9,10]. Sin embargo, no explican del todo los mecanismos fisiopatológicos de la enfermedad, debido a las características de patrón de herencia poligénica. Justamente allí es donde el conocimiento de la epigenética está permitiendo reunir elementos complementarios, que nos aproximan a una mayor comprensión de la enfermedad.
El análisis de cómo estos factores desencadenan una respuesta autoinmunitaria, relacionada con el inicio de la artritis reumatoide, ha derivado en un modelo preclínico que involucra al fenotipo de artritis reumatoide seropositiva, caracterizada por la elevación de biomarcadores, como el factor reumatoide y/o anticuerpos anti-péptido cíclico citrulinado, en fases previas a la enfermedad. Así, se sugiere que la interacción de los genes y el exposoma [11][12] podrían interactuar en un período previo a la aparición de los primeros síntomas articulares. No obstante, este modelo no es equiparable para los fenotipos seronegativos [13].
La epigenética constituye un fenómeno hereditario y reversible que compromete la expresión génica sin generar cambios en la secuencia primaria del ácido desoxirribonucleico (ADN), y sus tres modificaciones la representan:
-
La metilación del ADN que consiste en una adición del grupo metilo de S- adenosil metionina a una base de citosina en sitios CpG (sitios de ADN donde la citosina es seguida por una a nivel guanina).
-
La modificación de histonas mediante la metilación y acetilación; modificaciones químicas o de otro tipo de histonas, y modificaciones covalentes de proteínas no histonas.
-
La expresión desregulada de micro-ARN que actúan a nivel postranscripcional de los genes [14,15].
Estos mecanismos epigenéticos influyen en las consecuencias fenotípicas de la variación genética. Para explicar el mecanismo biológico de una patología, es necesario caracterizar la relación entre los efectos genéticos y epigenéticos. Estas relaciones pueden describirse como mediación, en la que la variación genética influye en la metilación, que luego influye en el fenotipo o interacción (también denominada modificación del efecto), en la que el efecto de la metilación difiere según el genotipo, o ambos [16].
El objetivo de esta revisión es identificar cómo una desregulación epigenética específica puede ofrecer una mayor comprensión de la fisiopatología de la enfermedad y tener un efecto en la prevención, diagnóstico e intervenciones terapéuticas.
Materiales y métodos
Estrategia de búsqueda y selección de estudios
Se utilizaron las bases de datos MEDLINE/PubMed, LILACS y Cochrane. Para ampliarlas, se completó con Google Académico y búsqueda manual. Se utilizaron los siguientes términos: "Rheumatoid Arthitis" [Mesh] AND "Epigenetic” [Mesh], AND “DNA methylation. Los criterios de inclusión para esta revisión fueron:
-
Trabajos publicados en idioma inglés y español.
-
Ensayos clínicos, estudios transversales, estudios de casos y controles, revisiones sistemáticas y metanálisis que incluyeron pacientes adultos con artritis reumatoide según los criterios de clasificación del American College of Rheumatology and European League Against Rheumatism (ACR/EULAR).
-
Trabajos publicados desde 2018 a 2022.
Los criterios de exclusión fueron:
-
Estudios sin relación con el tema.
-
Editoriales, revisiones narrativas, resúmenes de congresos;
-
Artículos sin acceso.
-
Datos insuficientes.
-
Publicaciones duplicadas.
Perspectivas de la epigenética en artritis reumatoide
La etiología de la artritis reumatoide, como en otras enfermedades autoinmunitarias, es multifactorial, jugando un papel importante los factores genéticos, ambientales y endógenos. Todos estos factores pueden modificar el paisaje epigenético de las células que son relevantes en la patogenia de la artritis reumatoide. Esto sugiere que la epigenética puede ser una interfaz común, a través de la cual los factores de riesgo, individualmente o en conjunto, actúan en el desarrollo de enfermedad. Numerosos estudios demuestran diferencias en el epigenoma de células relevantes en pacientes con artritis reumatoide en comparación con individuos sanos. El medio inflamatorio crónico en la artritis reumatoide puede afectar profundamente la epigenética de las células y podría invocar cambios sostenidos en los fenotipos celulares, obstruyendo la resolución de la artritis reumatoide y potenciando su cronicidad. La elucidación de cómo los factores de riesgo de la artritis reumatoide cambian su epigenética y cómo las alteraciones epigenéticas relacionadas con la enfermedad modifican la biología de la artritis reumatoide, es el siguiente paso hacia una mejor comprensión de esta patología [17].
Los mecanismos epigenéticos, como la metilación del ADN, la remodelación de la cromatina y los ácidos ribonucleicos (ARN) no codificantes; se han identificado como reguladores cruciales en la inmunidad celular. Esto, debido a sus mecanismos para modular la expresión génica y la transcripción en células y tejidos específicos. La metilación del ADN es una de las marcas epigenéticas más estudiadas e influye en una gran variedad de procesos biológicos, como la represión transcripcional, el silenciamiento reversible del promotor y la inestabilidad cromosómica. Una de las funciones cruciales de la metilación del ADN es el mantenimiento de la regulación de las células T. Por lo tanto, la metilación del ADN juega un papel crítico en numerosas enfermedades autoinmunes al alterar los perfiles de expresión génica. Los mecanismos epigenéticos involucrados en la artritis reumatoide incluyen estados de metilación alterados en las células T y B, así como en los fibroblastos sinoviales [18].
Varias líneas de evidencia sugieren que los cambios epigenéticos, incluida la metilación y los cambios en los microARN y ARN largos no codificantes, ocurren antes del tratamiento o son inducidos por el tratamiento y/o el envejecimiento, pudiendo tener un papel importante en los pacientes con artritis reumatoide refractaria inflamatoria persistente. La firma epigenética de la artritis reumatoide también puede cambiar como resultado del envejecimiento o factores ambientales. Además, los regímenes de tratamiento, que inicialmente son exitosos en pacientes con artritis reumatoide, pueden posteriormente fallar, sugiriendo un cambio en los mediadores de la enfermedad en estos individuos [19].
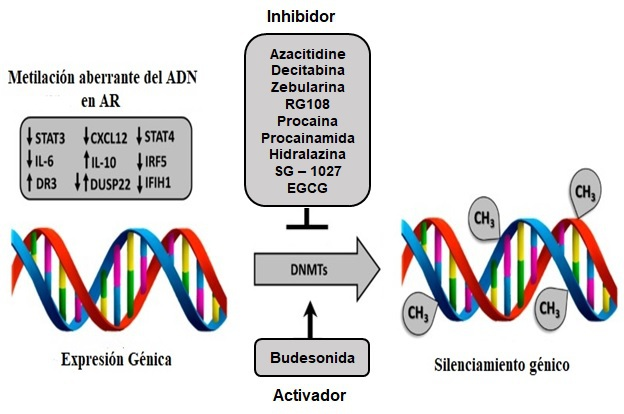
Algunos estudios han mostrado que la terapia con fármacos antirreumáticos modificadores de la enfermedad sintéticos convencionales, biológicos y/o corticosteroides pueden influenciar el estatus de la metilación del ADN. Dicha metilación es ejercida por una familia de ADN metiltransferasas, que son inhibidas o activadas por varios fármacos epigenéticos y en consecuencia modulan la expresión génica [20] (Figura 1).
Mecanismo potencial de genes diferencialmente metilados en la artritis reumatoide (AR) regulado por varios fármacos epigenéticos. La metilación del ADN es ejercida por ADN metiltransferasas. Las ADN metiltransferasas están influenciadas por varios fármacos epigenéticos que las inhiben o activan, lo que en consecuencia da como resultado la modulación de la expresión génica. La flecha hacia abajo indica genes hipometilados (STA3, interleucina-6, CXCL12, DUSP22, STAT4, IRF5, IFIH1) y la flecha hacia arriba indica genes hipermetilados (DR3, interleucina-10, DUSP22).
Además, se ha demostrado que los miARN juegan un papel importante en la artritis reumatoide. Entre ellos miR-146a, el cual se ha propuesto como un regulador clave del sistema inmunitario al controlar la secreción de citoquinas, la función de las células B y la señalización de factor nuclear-kβ, entre otros. Los niveles elevados de miR-146a se han asociado con la expresión de interleucina-17 en las células mononucleares de sangre periférica y la membrana sinovial de pacientes con artritis reumatoide. Otros microARN relevantes son miR-155, que produce la regulación positiva de factor de necrosis tumoral-α e interleucina-1β en las células mononucleares de sangre periférica de individuos con artritis reumatoide; miR-346 que controla la síntesis de factor de necrosis tumoral-β; miR-23b que suprime la inflamación autoinmune asociada a interleucina-17; y miR-126a que promueve la inhibición de ADN metiltransferasas, produciendo la hipometilación de CD70 en las células B y, por lo tanto, contribuyendo a la respuesta autoinmune [21].
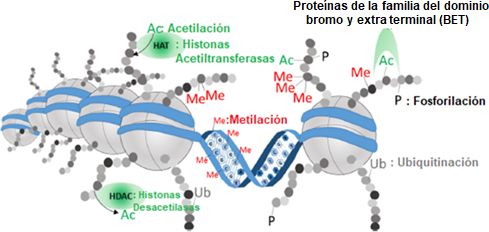
Finalmente, las enzimas modificadoras de histonas son reguladores epigenéticos implicados en el control de los procesos inflamatorios, incluida la activación, la supervivencia y la proliferación de células estromales e inmunitarias. Las histonas acetiltransferasas acetilan los residuos de lisina en las colas de las histonas, mientras que las histonas desacetilasas contrarrestan la actividad histonas acetiltransferasas al desacetilar las proteínas histonas. El delicado equilibrio entre el estado acetilado y el estado desacetilado de la cromatina afecta tanto la estructura global y el metabolismo de la cromatina generando la activación o represión génica. Además, las histonas acetiltransferasas y las histonas desacetilasas también pueden afectar el estado de acetilación de las proteínas que no son histonas, regulando así las proteínas de señalización y los factores de transcripción para influir en la expresión génica y la función celular como consecuencia, la función histonas desacetilasas podría ser esencial para el desarrollo y la perpetuación de enfermedades inflamatorias crónicas, como la artritis reumatoide [22] (Figura 2).
Modificaciones epigenéticas. El ADN (en azul) está enrollado alrededor de las histonas (gris). Las modificaciones en los aminoácidos de las colas de histonas que sobresalen pueden influir en la densidad del ADN y pueden reclutar complejos represores o transcripcionales. Las marcas de histonas son colocadas, leídas y eliminadas por enzimas especializadas. Como ejemplo, se muestran las histonas acetiltransferasas (HAT) y las histonas desacetilasas (HDAC). Las proteínas de la familia del dominio bromo y extra-terminal (BET) se unen a la lisina acetilada en las colas de las histonas, luego pueden promover la transcripción y el reclutamiento de enzimas epigenéticas para estabilizar aún más el estado de la cromatina abierta en un locus específico. Ac: acetilación, Me: metilación, P: fosforilación, Ub: ubiquitinación.
La epigenética en la artritis reumatoide
La epigenética representa un área de importancia para comprender algunos factores que contribuyen al riesgo de desarrollar artritis reumatoide, lo cual ha quedado en evidencia con los resultados de algunos estudios sobre como las modificaciones epigenéticas pueden afectar la expresión génica en esta entidad nosológica.
Tseng C et al., realizaron un estudio en una cohorte de pacientes con artritis reumatoide y controles sanos sobre el efecto de la metilación del ADN codificante de la molécula de muerte celular programada 1, observándose en los pacientes con artritis reumatoide, tratados con fármacos antirreumáticos modificadores de la enfermedad sintéticos convencionales , un aumento en la expresión de la molécula de muerte celular programada 1 y la hipometilación de dicha molécula en comparación a los controles. Además, la expresión de la molécula de muerte celular programada 1 es afectada en células portadoras del alelo T del polimorfismo rs41386349 en comparación con las células portadoras del alelo C. Esta expresión defectuosa de la molécula de muerte celular programada 1 puede conducir a una activación aberrante de las respuestas inmunitarias humorales y un aumento de autoanticuerpos. En consecuencia, esto implica un aumento de la seropositividad (factor reumatoide) [24].
Un estudio transversal demostró que la peptidilarginina deiminasa contribuye a la patogénesis de la artritis reumatoide al inhibir la apoptosis de los sinoviocitos similares a fibroblastos. La expresión disminuida de peptidilarginina deiminasa incrementa la apoptosis de los sinoviocitos similares a fibroblastos de pacientes con artritis reumatoide, mientras que la regulación positiva de peptidilarginina deiminasa podría disminuir la apoptosis de los sinoviocitos similares a fibroblastos, al interrumpir las modificaciones del residuo de arginina de la histona H3, que suprime la transcripción p21, proporcionando una nueva visión del papel anti-apoptótico de peptidilarginina deiminasa en el desarrollo de la artritis reumatoide y sus implicaciones en los mecanismos epigenéticos [25].
Ham S et al. evaluaron los perfiles de metilación del ADN de sinoviocitos aislados en cirugía de reemplazo articular de pacientes con artritis reumatoide y osteoartrosis. Entre los 28 micro ARN expresados diferencialmente (DEmiRs) entre artritis reumatoide y osteoartrosis, 27 micro ARN mostraron correlaciones significativas con la metilación del ADN a nivel de CpG, sugiriendo que los CpG individuales pueden tener un papel potencial en la regulación de los micro ARN [26].
En un metanálisis, en el que se incluyeron 22 estudios de casos y controles, (n = 10 489), se analizó si los polimorfismos de los genes microARN estaban asociados con la artritis reumatoide. En este metanálisis no se encontró la asociación del polimorfismo rs2910164 (G/C) miR-146a con artritis reumatoide, pero sí con el polimorfismo rs3746444 (T/C) miR-499, especialmente en poblaciones caucásicas [27].
Payet M et al., realizaron un metanálisis con el objetivo de evaluar la expresión de micro ARN, la metilación del ADN y las modificaciones de histonas en la artritis reumatoide, observando que en muestras humanas las expresiones de miR-155, miR-146a y miR-150 disminuyeron, mientras que la expresión de miR-410-3p aumentó, miR-146a disminuyó la expresión de la citoquina interleucina-17, y evidenciaron que el ADN parece estar hipometilado [28].
Mecanismos epigenéticos como biomarcadores
La complejidad poligénica, y las diferencias fenotípicas que presentan los pacientes con artritis reumatoide, impulsan la necesidad de abordar otras áreas que contribuyan a prevenir la aparición de esta patología. Asimismo, es necesario coadyuvar para un mejor control clínico de la enfermedad, para lo cual citamos los siguientes estudios que analizan algunos biomarcadores de importancia.
En un estudio, que incluyó un total de 17 muestras de tejido sinovial de tres cohortes, se evaluaron las diferencias a nivel de metilación del ADN y expresión génica y su relación con la patogénesis de la artritis reumatoide. La primera cohorte incluyó individuos que tenían artralgia y/o antecedentes familiares positivos para la artritis reumatoide, pero sin artritis, y que eran positivos para el factor reumatoideo inmunoglobulina M (IgM-RF) y/o anticuerpos contra la proteína anticitrulinada. La segunda cohorte consistió en individuos con artritis temprana, con una duración de la enfermedad menor a un año y que no habían recibido tratamiento previo con fármacos antirreumáticos modificadores de la enfermedad. La tercera cohorte incluyó pacientes con artritis reumatoide establecida, con una duración de la enfermedad de más de un año, con al menos una articulación inflamada y tratamiento activo. El análisis mostró que la sinovia de los pacientes con un mayor número de articulaciones inflamadas (igual o mayor a 9) era diferente a nivel de metilación del ADN y la expresión génica con respecto a la sinovia de los pacientes con un menor número de articulaciones inflamadas. La diferencia más significativa se encontró en la metilación de las regiones promotoras de MIRLET7B y MIR10B. A nivel de transcripción, fueron CCL13 y CLEC10A. También se evidenció metilación diferencial en las proximidades del promotor para CCL13, CXCL11 y CXCL9. Por último, se destaca la expresión diferencial de la transcripción, pertenecientes a ITGB2 y LCP2, con múltiples regiones de metilación diferencial observadas alrededor de ambos genes. Estos resultados apoyan el papel de los procesos epigenéticos/transcripcionales con un mayor compromiso articular [29].
Bae SC et al. evaluaron, a través de un metanálisis, la relación entre los niveles de miR-146a y la artritis reumatoide y la correlación con la actividad de la enfermedad. El análisis mostró que los niveles de miR-146a en el tejido/líquido sinovial fueron significativamente más altos en el grupo de pacientes con artritis reumatoide con respecto al grupo con osteoartrosis. Además, observaron una correlación positiva entre miR-146a y el nivel de la velocidad de sedimentación globular [30].
Ayeldeen G et al., en un estudio de casos y controles, en el cual evaluaron el uso de la expresión de micro ARN-146a y micro ARN-499 en el diagnóstico de pacientes con artritis reumatoide, demostraron que los polimorfismos de micro ARN-146a (rs2710164) y micro ARN-499 (rs3746444) se asociaban con mayor susceptibilidad para dicha enfermedad [31].
En otro estudio de casos y controles se evaluó la metilación del gen FCER1G y los miembros de la familia miR-17 como marcadores epigenéticos asociados con la artritis reumatoide, la actividad de la enfermedad y la expresión de interleucina-6. Los hallazgos mostraron una menor metilación de FCER1G y miR-106b en los pacientes con artritis reumatoide con respecto a los controles, pero no entre los grupos de pacientes con artritis reumatoide de alta actividad y remisión de la enfermedad. Además, observaron una correlación entre los niveles de interleucina-6 y la metilación de FCER1G [32].
Zhang LL et al. analizaron la asociación entre los polimorfismos de un solo nucleótido en el gen microRNA-146a (miR-146a) y la susceptibilidad a artritis reumatoide. El análisis demostró que el alelo T del polimorfismo de un solo nucleótido funcional rs2431697 aumenta el riesgo de artritis reumatoide y el tamaño del efecto del polimorfismo de nucleótido único es mayor en asiáticos que en europeos. Finalmente, hallaron una heterogeneidad significativa en el metanálisis trans-étnico, pero no en el metanálisis asiático [33].
En la determinación de los niveles de metilación de los genes ADAMTSL2 y LRPAP1 en el suero de pacientes con artritis reumatoide divididos según la actividad de la enfermedad y en comparación con el grupo control, se observó hipometilación del ADAMTSL2 en el grupo con DAS-28 (Disease Activity Score-28) con alta actividad de la enfermedad en comparación con los pacientes en remisión, así como en el grupo de control. Para el gen LRPAP1 hubo hipermetilación con DAS-28 en rango alto y en el grupo control en comparación con los pacientes en remisión [34].
La epigenética y terapéutica
El avance en la concepción sobre la medicina personalizada en reumatología constituye un reto para la identificación de biomarcadores que permitan identificar grupos de pacientes que puedan responder de manera diferente a los fármacos antirreumáticos sintéticos convencionales modificadores de la enfermedad y/o biológicos. Lo propio ocurre con la necesidad de desarrollar objetivos farmacológicos de tipo epigenético.
Gosselt HR et al. desarrollaron un ensayo clínico aleatorizado para analizar la asociación entre la hidroximetilación global del ADN, antes, durante y después de los tres meses de la terapia con metotrexato, y su relación con los cambios en la actividad de la enfermedad en pacientes con artritis reumatoide temprana. En dicho ensayo mostraron que una mayor metilación del ADN global inicial está asociada con la falta de respuesta clínica, determinada a los tres meses de tratamiento con metotrexato. Además, la metilación global media del ADN no cambió durante el tratamiento con este fármaco y la metilación global del ADN a los tres meses o más no se asoció con la eficacia clínica [35].
Nair et al., a través de un estudio observacional multicéntrico, investigaron la metilación diferencial del ADN como un biomarcador candidato de respuesta al metotrexato. Dicho estudio demostró que cuatro CpG (cg04334751, cg23700278, cg27427581, cg26764200) presentaron cambios en la metilación que predijeron una mejoría en articulaciones inflamadas, dolorosas, disminución de la proteína C reactiva y la escala DAS-28en pacientes con artritis reumatoide que iniciaron el tratamiento con metotrexato por primera vez, en control a los cuatro semanas y seis meses. Estos resultados representan un avance en la práctica actual al contribuir a una estrategia de medicina personalizada que puede permitir un cambio en la terapia a las cuatro semanas [36].
Kim KW et al. realizaron un estudio observacional donde trataron las células del estroma mesenquimal humano modificadas epigenéticamente (epi-hMSC), de pacientes con artritis reumatoide, con una combinación de agentes hipometilantes como 5-azacitidina o 5-aza-2′-desoxicitidina e inhibidores de histonas desacetilasas tipo tricostatina A o ácido valproico, probándose 36 combinaciones de tratamiento. Para ello, se utilizaron los niveles de expresión de indolamina 2,3-dioxigenasa e interleucina-10 en estroma mesenquimal humano tratadas durante 72 horas para seleccionar las combinaciones óptimas de fármacos. Se encontró que el tratamiento con la combinación de un agente hipometilante y un inhibidor de histonas desacetilasas aumentó las propiedades inmuno moduladoras de las estromas mesenquimal humanas, evidenciando que una combinación óptima de agentes hipometilantes e inhibidores de la histona desacetilasa puede mejorar el potencial inmunomodulador de las estromas mesenquimal humanas, lo que puede ser útil para el tratamiento de la artritis reumatoide [37].
Por otra parte, la cohorte de pacientes con artritis temprana de Rotterdam no identificó posiciones y regiones diferencialmente metiladas en el genoma en relación con los cambios en la escala de actividad medida con DAS-28 durante los primeros tres meses de tratamiento. Se requieren estudios más amplios para demostrar o descartar el uso de sitios de metilación del ADN como marcador predictivo para la respuesta a metotrexato [38].
Guderud K et al., en un estudio de cohorte y asociación de todo el epigenoma, el cual tuvo por objeto analizar las células T relevantes en artritis reumatoide (células T CD4+ vírgenes y de memoria) de dos cohortes de artritis reumatoide diferentes: pacientes con artritis reumatoide sin tratamiento previo con fármacos antirreumáticos modificadores de la enfermedad con enfermedad activa y pacientes con artritis reumatoide tratados con metotrexato que habían estado en remisión durante más de 12 meses, demostró más posiciones metiladas diferencialmente en la células T CD4+ de memoria que en las células T CD4+ vírgenes (904 frente a 19 posiciones metiladas diferencialmente) en pacientes con artritis reumatoide en comparación con los controles. La mayoría de las posiciones metiladas diferencialmente identificadas en pacientes con artritis reumatoide recién diagnosticados y sin tratamiento previo, con fármacos antirreumáticos modificadores de la enfermedad, con enfermedad activa mostraron un aumento de la metilación del ADN (39 posiciones metiladas diferencialmente), mientras que la mayoría de las posiciones metiladas diferencialmente identificadas en los pacientes con artritis reumatoide tratados con metotrexato en remisión mostraron una disminución de la metilación del ADN. Además, encontraron que aproximadamente un tercio de los 101 loci de riesgo de artritis reumatoide conocidos se superponían (±500 kb) con los posiciones metiladas diferencialmente. En particular, los intrones del gen UBASH3A albergan tanto el polimorfismos de un solo nucleótido de riesgo de artritis reumatoide como dos posiciones metiladas diferencialmente en las células T de memoria CD4+ [39].
Mortazavi S et al., realizaron un ensayo clínico aleatorizado para evaluar el fármaco β-d-mannurónico y su efecto sobre la expresión de miR-146a, sus dianas (IRAK1, TRAF6) y el factor de transcripción factor nuclear-κB en pacientes con artritis reumatoide. Después de 12 semanas de tratamiento con β-d-mannurónico observaron una disminución en la expresión génica de miR-146a, IRAK1, TRAF6, y factor nuclear-κB, así como en los niveles séricos de interleucina-6 y factor de necrosis tumoral-α [40].
Paoletti A et al., analizaron la expresión de factor de necrosis tumoral de membrana en monocitos sanguíneos, la polarización en macrófagos, la expresión de miR-155 y el efecto de anti-factor de necrosis tumoral en pacientes con artritis reumatoide. El análisis demostró que los pacientes con artritis reumatoide presentaban anomalías específicas de monocitos/macrófagos, que el aumento de factor de necrosis tumoral de membrana en los monocitos se asociaba con la actividad de la enfermedad, y que la maduración de los monocitos en macrófagos antiinflamatorios similares a M2 era deficiente. Por otra parte, el incremento de la expresión de miR-155 en monocitos/macrófagos podría ser responsable de este defecto de polarización de los monocitos, que podría ser parcialmente revertido por un anticuerpo monoclonal anti-factor de necrosis tumoral, y no por el receptor soluble. Por lo tanto, se puede sugerir que un antagonista del miR-155 en monocitos/macrófagos puede representar una nueva estrategia terapéutica en la artritis reumatoide [41].
Además, en dos ensayos clínicos aleatorizados demostraron el efecto de los fármacos en la expresión de microARNs. Un primer ensayo comprobó, en pacientes con artritis reumatoide temprana, que los niveles elevados de miR-27a-3p estaban asociado con remisión después de 12 meses de tratamiento con adalimumab en combinación con metotrexato. En este mismo ensayo demostraron que el miR-16-5p y el miR-22-3p pueden ser biomarcadores de actividad de la enfermedad y predictores de respuesta al tratamiento con metotrexato [42]. El segundo ensayo demostró que pacientes tratados con el medicamento β-d-mannurónico presentaban una disminución del miR-155, aumento del SOCS1, SHIP1, y disminución del factor de transcripción factor nuclear-κB, generando disminución de la actividad de la enfermedad cuantificada por la escala del DAS 28 y mejoría en la calidad de vida medido por la escala HAQ [43].
En una revisión sistemática sobre la utilidad de los micro ARN como biomarcadores de respuesta al tratamiento con metotrexato, inhibidores del factor de necrosis tumoral (anti-TNF) y rituximab en los pacientes con artritis reumatoide, sugiriendo que los micro ARN más polivalentes fueron miR-146a, que predijo la respuesta al metotrexato e inhibidores del factor de necrosis tumoral y miR-125b, que predice la respuesta a infliximab y rituximab [44].
Discusión
En artritis reumatoide se pudo determinar que los microARN más relevantes como factores de susceptibilidad los representaron el micro ARN-146a y micro ARN-499, siendo importante analizar el papel de estas moléculas en diferentes poblaciones étnicas, y probablemente correlacionarlas con biomarcadores serológicos y clínicos. También fueron relacionadas con la actividad de la enfermedad, y como posibles predictores de respuesta al tratamiento.
Se ha evidenciado la acción del metotrexato sobre la hidroximetilación en los pacientes con artritis reumatoide, siendo importante plantearse trabajos de investigación evaluando el efecto de las diferentes dosis del medicamento sobre la metilación. Hemos observado la descripción de fármacos experimentales, como el β-d-mannurónico, y otros que intervienen en diferentes procesos relacionados con la epigenética. Sin embargo, el soporte científico actual para su empleo en seres humanos es incompleto, requiriéndose estudios que aborden el impacto terapéutico en los mecanismos epigenéticos de los pacientes con artritis reumatoide.
Pudimos revisar que existen estudios no concluyentes sobre ciertos mecanismos epigenéticos, lo cual corrobora la complejidad que subyace en el entendimiento de la artritis reumatoide. Esto, debido a que se trata de una enfermedad crónica, poligénica, que requiere para su expresión clínica la interacción con el exposoma, entendiéndolo como la conjunción de exposición externa especifica (contaminación ambiental, tabaco, etc.), la exposición externa general (estatus socioeconómico, educación, etc.), y exposición interna (inflamación, estrés oxidativo, microbioma, etc.) [45]. Como correlación de este último elemento, es menester señalar cómo estos efectos del ambiente externo inducen en los organismos cambios en el estado redox celular y, aún más específicamente, en su compartimiento redox nuclear. Todo ello cual induciría en la modulación de la regulación epigenética en las células, mediante la modificación postraduccional de estas proteínas como la nitrosilación de histonas, la carbonilación o la glutationilación [46]. Por lo tanto, las futuras iniciativas de investigación deben incorporar estos elementos para su análisis más integrador.
Conclusiones
La epigenética representa un componente dentro de la complejidad fisiopatológica de la artritis reumatoide, en su prevención, diagnóstico y terapéutica. En consecuencia, su avance y relación con los otros factores integradores de la enfermedad permitirá una mejor comprensión de esta patología.

